12 Tidy data
12.1 Introduzione
“Le famiglie felici sono tutte uguali; ogni famiglia infelice è infelice a modo suo”. — Leo Tolstoy
“Le serie di dati ordinate sono tutte uguali, ma ogni serie di dati disordinata è disordinata a modo suo”. — Hadley Wickham
In questo capitolo, imparerete un modo coerente per organizzare i vostri dati in R, un’organizzazione chiamata tidy data. Mettere i vostri dati in questo formato richiede un po’ di lavoro iniziale, ma questo lavoro ripaga nel lungo termine. Una volta che avete dati ordinati e gli strumenti ordinati forniti dai pacchetti del tidyverse, passerete molto meno tempo a mescolare i dati da una rappresentazione all’altra, permettendovi di dedicare più tempo alle domande analitiche che avete a disposizione.
Questo capitolo vi darà un’introduzione pratica ai ‘tidy data’(dati ordinati, organizzati) e agli strumenti di accompagnamento nel pacchetto tidyr. Se volete saperne di più sulla teoria sottostante, potreste apprezzare il documento Tidy Data pubblicato nel Journal of Statistical Software, http://www.jstatsoft.org/v59/i10/paper.
12.2 Tidy data
È possibile rappresentare gli stessi dati sottostanti in più modi. L’esempio qui sotto mostra gli stessi dati organizzati in quattro modi diversi. Ogni set di dati mostra gli stessi valori di quattro variabili paese, anno, popolazione, e casi, ma ogni set di dati organizza i valori in un modo diverso.
table1
#> # A tibble: 6 × 4
#> country year cases population
#> <chr> <dbl> <dbl> <dbl>
#> 1 Afghanistan 1999 745 19987071
#> 2 Afghanistan 2000 2666 20595360
#> 3 Brazil 1999 37737 172006362
#> 4 Brazil 2000 80488 174504898
#> 5 China 1999 212258 1272915272
#> 6 China 2000 213766 1280428583
table2
#> # A tibble: 12 × 4
#> country year type count
#> <chr> <dbl> <chr> <dbl>
#> 1 Afghanistan 1999 cases 745
#> 2 Afghanistan 1999 population 19987071
#> 3 Afghanistan 2000 cases 2666
#> 4 Afghanistan 2000 population 20595360
#> 5 Brazil 1999 cases 37737
#> 6 Brazil 1999 population 172006362
#> # … with 6 more rows
table3
#> # A tibble: 6 × 3
#> country year rate
#> <chr> <dbl> <chr>
#> 1 Afghanistan 1999 745/19987071
#> 2 Afghanistan 2000 2666/20595360
#> 3 Brazil 1999 37737/172006362
#> 4 Brazil 2000 80488/174504898
#> 5 China 1999 212258/1272915272
#> 6 China 2000 213766/1280428583
# Distribuito su due tibble
table4a # casi
#> # A tibble: 3 × 3
#> country `1999` `2000`
#> <chr> <dbl> <dbl>
#> 1 Afghanistan 745 2666
#> 2 Brazil 37737 80488
#> 3 China 212258 213766
table4b # popolazione
#> # A tibble: 3 × 3
#> country `1999` `2000`
#> <chr> <dbl> <dbl>
#> 1 Afghanistan 19987071 20595360
#> 2 Brazil 172006362 174504898
#> 3 China 1272915272 1280428583Sono tutte rappresentazioni degli stessi dati sottostanti, ma non sono ugualmente facili da usare. Un dataset, il dataset ordinato, sarà molto più facile da lavorare all’interno del tidyverse.
Ci sono tre regole correlate che rendono un dataset ordinato:
- Ogni variabile deve avere la propria colonna.
- Ogni osservazione deve avere la sua riga.
- Ogni valore deve avere la propria cella.
La figura 12.1 mostra visivamente le regole.

Figure 12.1: Seguire tre regole rende un set di dati ordinato: le variabili sono nelle colonne, le osservazioni sono nelle righe e i valori sono nelle celle.
Queste tre regole sono interrelate perché è impossibile soddisfare solo due delle tre. Questa interrelazione porta ad un insieme ancora più semplice di istruzioni pratiche:
- Metti ogni set di dati in una tibble.
- Metti ogni variabile in una colonna.
In questo esempio, solo la table1 è ordinata. È l’unica rappresentazione in cui ogni colonna è una variabile.
Perché assicurarsi che i dati siano ordinati? Ci sono due vantaggi principali:
C’è un vantaggio generale nel scegliere un modo coerente di memorizzare dati. Se hai una struttura di dati coerente, è più facile imparare gli strumenti che lavorano con essa perché hanno un’uniformità di fondo.
C’è un vantaggio specifico nel mettere le variabili in colonne perché permette alla natura vettoriale di R di brillare. Come avete imparato in mutate e riassunti raggruppati, la maggior parte funzioni built-in di R lavorano con vettori di valori. Questo rende la trasformazione dati ordinati in modo particolarmente naturale.
dplyr, ggplot2 e tutti gli altri pacchetti del tidyverse sono progettati per lavorare con dati ordinati. Ecco un paio di piccoli esempi che mostrano come potreste lavorare con table1.
# Calcolare il tasso per 10.000
table1 %>%
mutate(rate = cases / population * 10000)
#> # A tibble: 6 × 5
#> country year cases population rate
#> <chr> <dbl> <dbl> <dbl> <dbl>
#> 1 Afghanistan 1999 745 19987071 0.373
#> 2 Afghanistan 2000 2666 20595360 1.29
#> 3 Brazil 1999 37737 172006362 2.19
#> 4 Brazil 2000 80488 174504898 4.61
#> 5 China 1999 212258 1272915272 1.67
#> 6 China 2000 213766 1280428583 1.67
# Calcolare i casi per anno
table1 %>%
count(year, wt = cases)
#> # A tibble: 2 × 2
#> year n
#> <dbl> <dbl>
#> 1 1999 250740
#> 2 2000 296920
# Visualizzare i cambiamenti nel tempo
library(ggplot2)
ggplot(table1, aes(year, cases)) +
geom_line(aes(group = country), colour = "grey50") +
geom_point(aes(colour = country))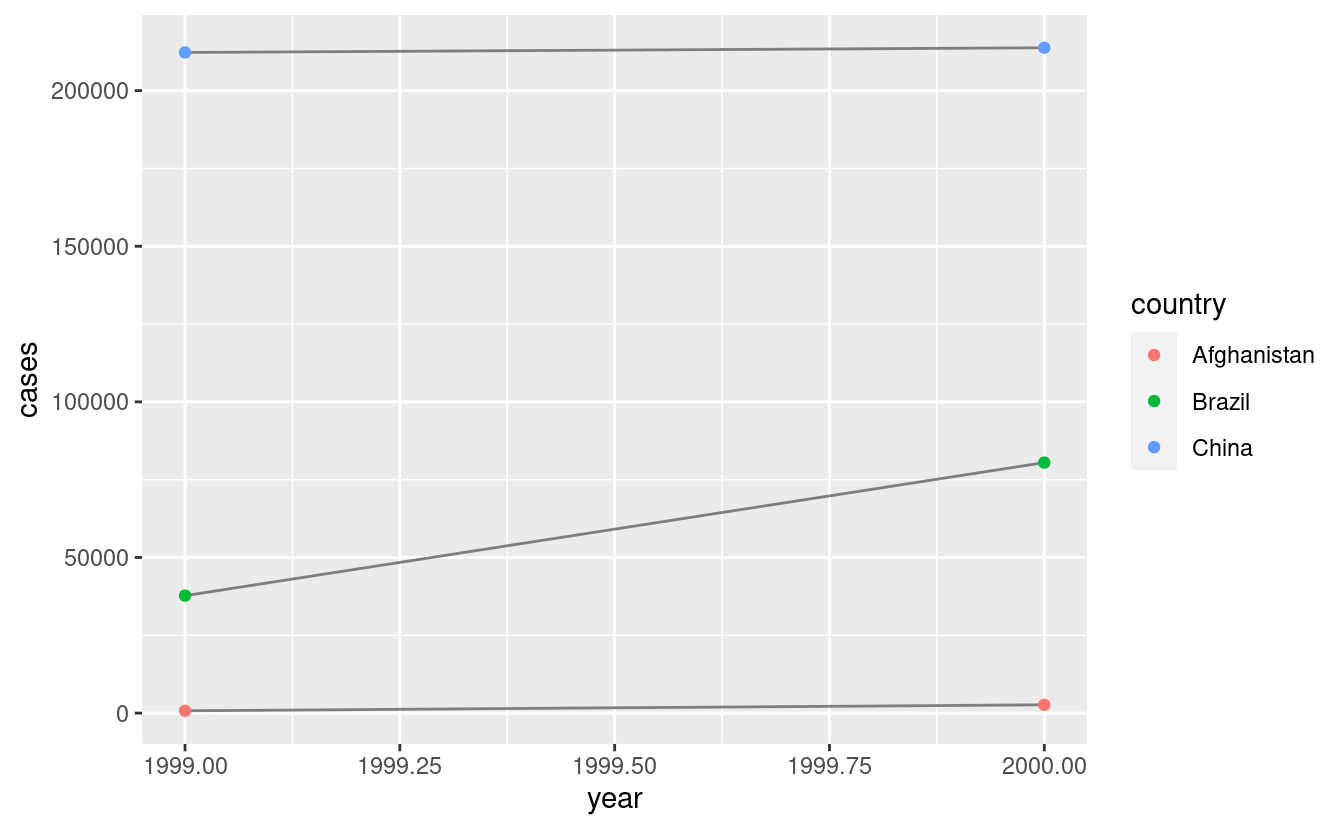
12.2.1 Esercizi
Usando la prosa, descrivete come sono organizzate le variabili e le osservazioni in ciascuna delle tabelle di esempio.
-
Calcola il
rateper latable2, e latable4a+table4b. Dovrai eseguire quattro operazioni:- Estrarre il numero di casi di TBC per paese e per anno.
- Estrarre la popolazione corrispondente per paese e per anno.
- Dividere i casi per la popolazione e moltiplicare per 10000.
- Memorizzare di nuovo nel posto appropriato.
Quale rappresentazione è più facile da lavorare? Qual è la più difficile? Perché?
Ricreate il grafico che mostra il cambiamento dei casi nel tempo usando la
tabella2 invece ditable1`. Cosa devi fare per prima cosa?
12.3 Pivoting
I principi dei tidy data sembrano così ovvi che potreste chiedervi se incontrerete mai un set di dati che non sia ordinato. Sfortunatamente, però, la maggior parte dei dati che incontrerete saranno disordinati. Ci sono due ragioni principali:
La maggior parte delle persone non ha familiarità con i principi dei dati ordinati, ed è difficile ricavarli da soli, a meno che non si passi molto tempo a lavorare con i dati.
I dati sono spesso organizzati per facilitare qualche uso diverso dall’analisi. Per esempio, i dati sono spesso organizzati per rendere l’inserimento il più facile possibile.
Questo significa che per la maggior parte delle analisi reali, dovrete fare un po’ di ordine. Il primo passo è sempre quello di capire quali sono le variabili e le osservazioni. A volte questo è facile; altre volte avrete bisogno di consultare le persone che hanno originariamente generato i dati. Il secondo passo è quello di risolvere uno dei due problemi comuni:
Una variabile potrebbe essere distribuita su più colonne.
Un’osservazione potrebbe essere sparsa su più righe.
Tipicamente un set di dati soffrirà solo di uno di questi problemi; soffrirà di entrambi solo se siete davvero sfortunati! Per risolvere questi problemi, avrete bisogno delle due funzioni più importanti di tidyr: pivot_longer() e pivot_wider().
12.3.1 Longer
Un problema comune è un dataset dove alcuni dei nomi delle colonne non sono nomi di variabili, ma valori di una variabile. Prendete la table4a: i nomi delle colonne 1999 e 2000 rappresentano i valori della variabile year, i valori nelle colonne 1999 e 2000 rappresentano i valori della variabile cases, e ogni riga rappresenta due osservazioni, non una.
table4a
#> # A tibble: 3 × 3
#> country `1999` `2000`
#> <chr> <dbl> <dbl>
#> 1 Afghanistan 745 2666
#> 2 Brazil 37737 80488
#> 3 China 212258 213766Per riordinare un set di dati come questo, abbiamo bisogno di pivot le colonne offensive in una nuova coppia di variabili. Per descrivere questa operazione abbiamo bisogno di tre parametri:
L’insieme delle colonne i cui nomi sono valori, non variabili. In questo esempio, queste sono le colonne
1999e2000.Il nome della variabile in cui spostare i nomi delle colonne. Qui è
year.Il nome della variabile in cui spostare i valori della colonna. Qui è
case.
Insieme questi parametri generano la chiamata a pivot_longer():
table4a %>%
pivot_longer(c(`1999`, `2000`), names_to = "year", values_to = "cases")
#> # A tibble: 6 × 3
#> country year cases
#> <chr> <chr> <dbl>
#> 1 Afghanistan 1999 745
#> 2 Afghanistan 2000 2666
#> 3 Brazil 1999 37737
#> 4 Brazil 2000 80488
#> 5 China 1999 212258
#> 6 China 2000 213766Le colonne per fare il pivot sono specificate con la notazione in stile dplyr::select(). Qui ci sono solo due colonne, quindi le elenchiamo singolarmente. Notate che “1999” e “2000” sono nomi non sintattici (perché non iniziano con una lettera), quindi dobbiamo circondarli di backtick. Per rinfrescarti la memoria sugli altri modi di selezionare le colonne, vedi select.
year e cases non esistono in table4a quindi mettiamo i loro nomi tra virgolette.

Figure 12.2: Pivot di table4 in forma ‘long’ e ordinata.
Nel risultato finale, le colonne pivotate vengono eliminate e si ottengono nuove colonne year e cases. Altrimenti, le relazioni tra le variabili originali sono conservate. Visivamente, questo è mostrato nella figura 12.2.
La funzione pivot_longer() rende i set di dati più lunghi aumentando il numero di righe e diminuendo il numero di colonne. Non credo che abbia senso descrivere un set di dati come se fosse in “forma lunga”. La lunghezza è un termine relativo, e si può solo dire (per esempio) che il dataset A è più lungo del dataset B.
Possiamo usare pivot_longer() per riordinare table4b in modo simile. L’unica differenza è la variabile memorizzata nei valori delle celle:
table4b %>%
pivot_longer(c(`1999`, `2000`), names_to = "year", values_to = "population")
#> # A tibble: 6 × 3
#> country year population
#> <chr> <chr> <dbl>
#> 1 Afghanistan 1999 19987071
#> 2 Afghanistan 2000 20595360
#> 3 Brazil 1999 172006362
#> 4 Brazil 2000 174504898
#> 5 China 1999 1272915272
#> 6 China 2000 1280428583Per combinare le versioni riordinate di table4a e table4b in una singola tibble, dobbiamo usare dplyr::left_join(), che imparerai in dati relazionali.
tidy4a <- table4a %>%
pivot_longer(c(`1999`, `2000`), names_to = "year", values_to = "cases")
tidy4b <- table4b %>%
pivot_longer(c(`1999`, `2000`), names_to = "year", values_to = "population")
left_join(tidy4a, tidy4b)
#> Joining with `by = join_by(country, year)`
#> # A tibble: 6 × 4
#> country year cases population
#> <chr> <chr> <dbl> <dbl>
#> 1 Afghanistan 1999 745 19987071
#> 2 Afghanistan 2000 2666 20595360
#> 3 Brazil 1999 37737 172006362
#> 4 Brazil 2000 80488 174504898
#> 5 China 1999 212258 1272915272
#> 6 China 2000 213766 128042858312.3.2 Wider
pivot_wider() è l’opposto di pivot_longer(). Si usa quando un’osservazione è sparsa su più righe. Per esempio, prendete la table2: un’osservazione è un paese in un anno, ma ogni osservazione è sparsa su due righe.
table2
#> # A tibble: 12 × 4
#> country year type count
#> <chr> <dbl> <chr> <dbl>
#> 1 Afghanistan 1999 cases 745
#> 2 Afghanistan 1999 population 19987071
#> 3 Afghanistan 2000 cases 2666
#> 4 Afghanistan 2000 population 20595360
#> 5 Brazil 1999 cases 37737
#> 6 Brazil 1999 population 172006362
#> # … with 6 more rowsPer riordinare il tutto, analizziamo prima la rappresentazione in modo simile a pivot_longer(). Questa volta, però, abbiamo bisogno solo di due parametri:
La colonna da cui prendere i nomi delle variabili. Qui è
type.La colonna da cui prendere i valori. Qui è
count.
Una volta capito questo, possiamo usare pivot_wider(), come mostrato programmaticamente qui sotto, e visivamente nella figura 12.3.
table2 %>%
pivot_wider(names_from = type, values_from = count)
#> # A tibble: 6 × 4
#> country year cases population
#> <chr> <dbl> <dbl> <dbl>
#> 1 Afghanistan 1999 745 19987071
#> 2 Afghanistan 2000 2666 20595360
#> 3 Brazil 1999 37737 172006362
#> 4 Brazil 2000 80488 174504898
#> 5 China 1999 212258 1272915272
#> 6 China 2000 213766 1280428583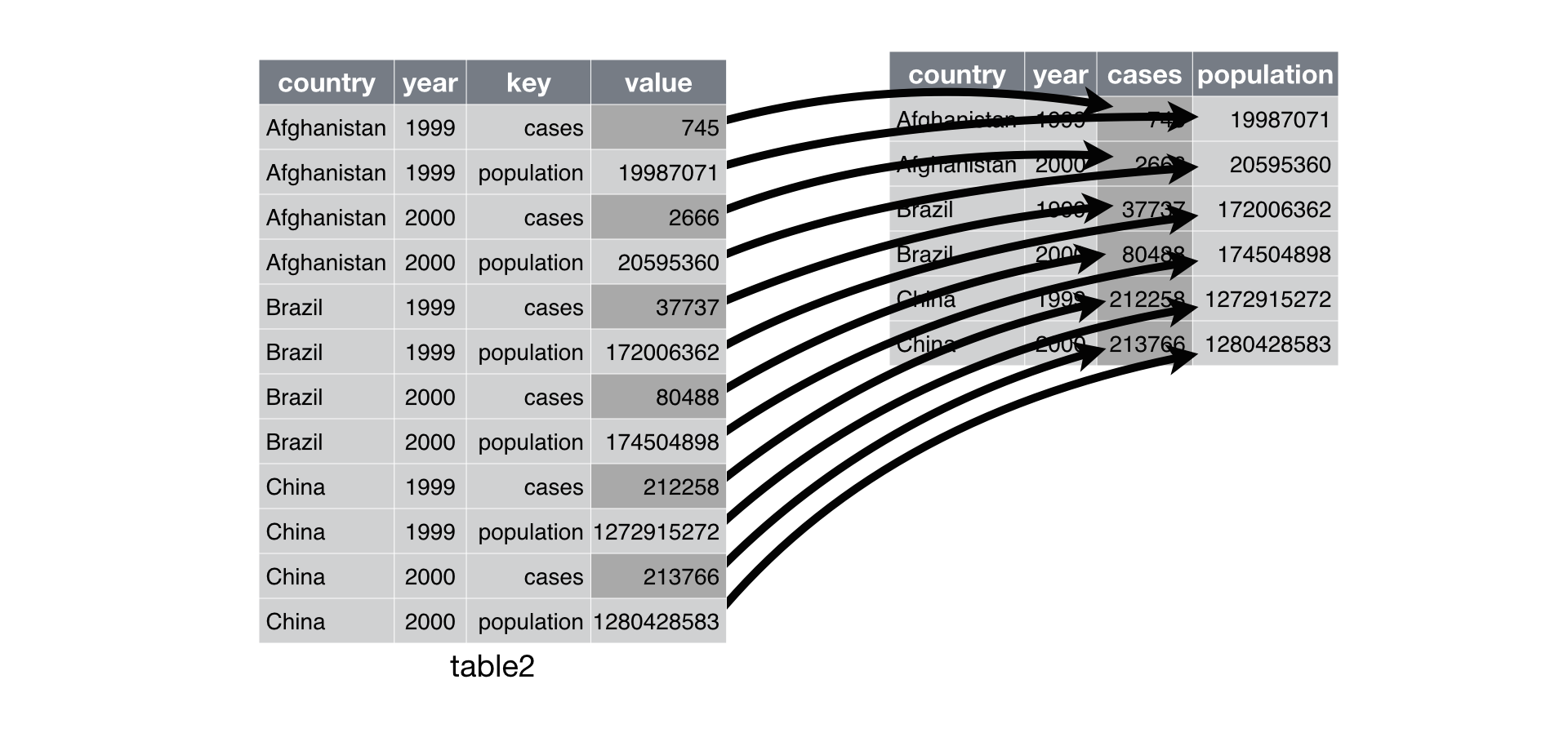
Figure 12.3: Pivoting table2 into a “wider”, tidy form.
Come avrete capito dai loro nomi, pivot_wider() e pivot_longer() sono complementari. pivot_longer() rende le tabelle larghe più strette e lunghe; pivot_wider() rende le tabelle lunghe più corte e larghe.
12.3.3 Esercizi
-
Perché i metodi
pivot_longer()epivot_wider()non sono perfettamente simmetrici?
Considerate attentamente il seguente esempio:stocks <- tibble( year = c(2015, 2015, 2016, 2016), half = c( 1, 2, 1, 2), return = c(1.88, 0.59, 0.92, 0.17) ) stocks %>% pivot_wider(names_from = year, values_from = return) %>% pivot_longer(`2015`:`2016`, names_to = "year", values_to = "return")(Suggerimento: guardate i tipi di variabili e pensate ai nomi delle colonne).
pivot_longer()ha un argomentonames_ptypes, per esempionames_ptypes = list(year = double()). Cosa fa? -
Perché questo codice fallisce?
table4a %>% pivot_longer(c(1999, 2000), names_to = "year", values_to = "cases") #> Error in `pivot_longer()`: #> ! Can't subset columns past the end. #> ℹ Locations 1999 and 2000 don't exist. #> ℹ There are only 3 columns. -
Cosa succederebbe se allargassi questa tabella? Perché? Come potresti aggiungere una nuova colonna per identificare in modo univoco ogni valore?
people <- tribble( ~name, ~names, ~values, #-----------------|--------|------ "Phillip Woods", "age", 45, "Phillip Woods", "height", 186, "Phillip Woods", "age", 50, "Jessica Cordero", "age", 37, "Jessica Cordero", "height", 156 ) -
Metti in ordine la semplice tibble qui sotto. Devi renderla più larga o più lunga, quali sono le variabili?
preg <- tribble( ~pregnant, ~male, ~female, "yes", NA, 10, "no", 20, 12 )
12.4 Separare e unire
Finora hai imparato come mettere in ordine table2 e table4, ma non table3. La table3 ha un problema diverso: abbiamo una colonna (rate) che contiene due variabili (casi e popolazione). Per risolvere questo problema, avremo bisogno della funzione separate(). Imparerete anche il complemento di separate(): unite(), che si usa se una singola variabile è distribuita su più colonne.
12.4.1 Separare
La funzione separate() separa una colonna in colonne multiple, dividendo ogni volta che appare un carattere separatore. Prendiamo ad esempio table3:
table3
#> # A tibble: 6 × 3
#> country year rate
#> <chr> <dbl> <chr>
#> 1 Afghanistan 1999 745/19987071
#> 2 Afghanistan 2000 2666/20595360
#> 3 Brazil 1999 37737/172006362
#> 4 Brazil 2000 80488/174504898
#> 5 China 1999 212258/1272915272
#> 6 China 2000 213766/1280428583La colonna rate contiene entrambe le variabili cases e population, e abbiamo bisogno di dividerla in due variabili. La funzione separate() prende il nome della colonna da separare e i nomi delle colonne in cui separare, come mostrato nella figura 12.4 e nel codice sottostante.
table3 %>%
separate(rate, into = c("cases", "population"))
#> # A tibble: 6 × 4
#> country year cases population
#> <chr> <dbl> <chr> <chr>
#> 1 Afghanistan 1999 745 19987071
#> 2 Afghanistan 2000 2666 20595360
#> 3 Brazil 1999 37737 172006362
#> 4 Brazil 2000 80488 174504898
#> 5 China 1999 212258 1272915272
#> 6 China 2000 213766 1280428583
Figure 12.4: Separating table3 makes it tidy
Per default, separate() dividerà i valori ovunque veda un carattere non alfanumerico (cioè un carattere che non sia un numero o una lettera). Per esempio, nel codice qui sopra, separate() divide i valori di rate in corrispondenza dei caratteri ‘forward slash’. Se volete usare un carattere specifico per separare una colonna, potete passare il carattere all’argomento sep di separate(). Per esempio, potremmo riscrivere il codice sopra come:
(Formalmente, sep è un’espressione regolare, di cui imparerai di più in stringhe).
Guardate attentamente i tipi di colonna: noterete che cases e population sono colonne di caratteri. Questo è il comportamento predefinito di separate(): lascia il tipo di colonna così com’è. Qui, tuttavia, non è molto utile, dato che sono davvero numeri. Possiamo chiedere a separate() di provare a convertire in tipi migliori usando convert = TRUE.:
table3 %>%
separate(rate, into = c("cases", "population"), convert = TRUE)
#> # A tibble: 6 × 4
#> country year cases population
#> <chr> <dbl> <int> <int>
#> 1 Afghanistan 1999 745 19987071
#> 2 Afghanistan 2000 2666 20595360
#> 3 Brazil 1999 37737 172006362
#> 4 Brazil 2000 80488 174504898
#> 5 China 1999 212258 1272915272
#> 6 China 2000 213766 1280428583Potete anche passare un vettore di interi a sep. separate() interpreterà gli interi come posizioni in cui dividere. I valori positivi partono da 1 all’estrema sinistra delle stringhe; i valori negativi partono da -1 all’estrema destra delle stringhe. Quando si usano gli interi per separare le stringhe, la lunghezza di sep dovrebbe essere inferiore al numero di nomi in into.
Puoi usare questa disposizione per separare le ultime due cifre di ogni anno. Questo rende questi dati meno ordinati, ma è utile in altri casi, come vedrai tra poco.
table3 %>%
separate(year, into = c("century", "year"), sep = 2)
#> # A tibble: 6 × 4
#> country century year rate
#> <chr> <chr> <chr> <chr>
#> 1 Afghanistan 19 99 745/19987071
#> 2 Afghanistan 20 00 2666/20595360
#> 3 Brazil 19 99 37737/172006362
#> 4 Brazil 20 00 80488/174504898
#> 5 China 19 99 212258/1272915272
#> 6 China 20 00 213766/128042858312.4.2 Unite
unite()è l’inverso di separate(): combina colonne multiple in una singola colonna. Ti servirà molto meno frequentemente di separate(), ma è comunque uno strumento utile da avere in tasca.

Figure 12.5: Uniting table5 makes it tidy
Possiamo usare unite() per riunire le colonne century e year che abbiamo creato nell’ultimo esempio. Questi dati vengono salvati come tidyr::table5. unite() prende un data frame, il nome della nuova variabile da creare e un insieme di colonne da combinare, sempre specificate in stile dplyr::select():
table5 %>%
unite(new, century, year)
#> # A tibble: 6 × 3
#> country new rate
#> <chr> <chr> <chr>
#> 1 Afghanistan 19_99 745/19987071
#> 2 Afghanistan 20_00 2666/20595360
#> 3 Brazil 19_99 37737/172006362
#> 4 Brazil 20_00 80488/174504898
#> 5 China 19_99 212258/1272915272
#> 6 China 20_00 213766/1280428583In questo caso dobbiamo anche usare l’argomento sep. Il default metterà un underscore (_) tra i valori delle diverse colonne. Qui non vogliamo nessun separatore quindi usiamo "":
table5 %>%
unite(new, century, year, sep = "")
#> # A tibble: 6 × 3
#> country new rate
#> <chr> <chr> <chr>
#> 1 Afghanistan 1999 745/19987071
#> 2 Afghanistan 2000 2666/20595360
#> 3 Brazil 1999 37737/172006362
#> 4 Brazil 2000 80488/174504898
#> 5 China 1999 212258/1272915272
#> 6 China 2000 213766/128042858312.4.3 Esercizi
-
Cosa fanno gli argomenti
extraefillinseparate()? Sperimentate le varie opzioni per i seguenti due set di dati giocattolo. Sia
unite()cheseparate()hanno un argomentoremove. Che cosa fa fa? Perché dovreste impostarlo aFALSE?Confrontate e contrapponete
separate()eextract(). Perché ci sono tre varianti di separazione (per posizione, per separatore e con gruppi), ma solo una unite?
12.5 Valori mancanti
Cambiare la rappresentazione di un set di dati fa emergere un’importante sottigliezza sui valori mancanti. Sorprendentemente, un valore può essere mancante in uno dei due modi possibili:
-
Esplicitamente, cioè contrassegnato da
NA. - Implicitamente, cioè semplicemente non presente nei dati.
Illustriamo questa idea con un insieme di dati molto semplice:
stocks <- tibble(
year = c(2015, 2015, 2015, 2015, 2016, 2016, 2016),
qtr = c( 1, 2, 3, 4, 2, 3, 4),
return = c(1.88, 0.59, 0.35, NA, 0.92, 0.17, 2.66)
)Ci sono due valori mancanti in questo set di dati:
Il rendimento per il quarto trimestre del 2015 è esplicitamente mancante, perché la cella dove dovrebbe essere il suo valore contiene invece
NA.Il rendimento per il primo trimestre del 2016 è implicitamente mancante, perché semplicemente non appare nel set di dati.
Un modo per pensare alla differenza è con questo koan zen: Un valore mancante esplicito è la presenza di un’assenza; un valore mancante implicito è l’assenza di una presenza.
Il modo in cui un set di dati è rappresentato può rendere espliciti i valori impliciti. Per esempio, possiamo rendere esplicito il valore mancante implicito mettendo gli anni nelle colonne:
stocks %>%
pivot_wider(names_from = year, values_from = return)
#> # A tibble: 4 × 3
#> qtr `2015` `2016`
#> <dbl> <dbl> <dbl>
#> 1 1 1.88 NA
#> 2 2 0.59 0.92
#> 3 3 0.35 0.17
#> 4 4 NA 2.66Poiché questi valori mancanti espliciti potrebbero non essere importanti in altre rappresentazioni dei dati, potete impostare values_drop_na = TRUE in pivot_longer() per rendere impliciti i valori mancanti espliciti:
stocks %>%
pivot_wider(names_from = year, values_from = return) %>%
pivot_longer(
cols = c(`2015`, `2016`),
names_to = "year",
values_to = "return",
values_drop_na = TRUE
)
#> # A tibble: 6 × 3
#> qtr year return
#> <dbl> <chr> <dbl>
#> 1 1 2015 1.88
#> 2 2 2015 0.59
#> 3 2 2016 0.92
#> 4 3 2015 0.35
#> 5 3 2016 0.17
#> 6 4 2016 2.66Un altro importante strumento per rendere espliciti i valori mancanti nei dati ordinati è complete():
stocks %>%
complete(year, qtr)
#> # A tibble: 8 × 3
#> year qtr return
#> <dbl> <dbl> <dbl>
#> 1 2015 1 1.88
#> 2 2015 2 0.59
#> 3 2015 3 0.35
#> 4 2015 4 NA
#> 5 2016 1 NA
#> 6 2016 2 0.92
#> # … with 2 more rowscomplete() prende un insieme di colonne e trova tutte le combinazioni uniche. Poi si assicura che il dataset originale contenga tutti quei valori, riempiendo i NA espliciti dove necessario.
C’è un altro importante strumento che dovreste conoscere per lavorare con i valori mancanti. A volte, quando una fonte di dati è stata usata principalmente per l’inserimento di dati, i valori mancanti indicano che il valore precedente dovrebbe essere portato avanti:
treatment <- tribble(
~ person, ~ treatment, ~response,
"Derrick Whitmore", 1, 7,
NA, 2, 10,
NA, 3, 9,
"Katherine Burke", 1, 4
)Potete riempire questi valori mancanti con fill(). Prende un insieme di colonne in cui volete che i valori mancanti siano sostituiti dal più recente valore non mancante (a volte chiamato ultima osservazione portata avanti).
treatment %>%
fill(person)
#> # A tibble: 4 × 3
#> person treatment response
#> <chr> <dbl> <dbl>
#> 1 Derrick Whitmore 1 7
#> 2 Derrick Whitmore 2 10
#> 3 Derrick Whitmore 3 9
#> 4 Katherine Burke 1 412.5.1 Esercizi
Confrontate e contrapponete gli argomenti di
fillapivot_wider()ecomplete().Cosa fa l’argomento direction di
fill()?
12.6 Caso di studio
Per finire il capitolo, mettiamo insieme tutto quello che abbiamo imparato per affrontare un realistico problema di riordino dei dati. Il dataset tidyr::who contiene casi di tubercolosi (TB) suddivisi per anno, paese, età, sesso e metodo di diagnosi. I dati provengono dal 2014 World Health Organization Global Tuberculosis Report, disponibile su http://www.who.int/tb/country/data/download/en/.
C’è una ricchezza di informazioni epidemiologiche in questo set di dati, ma è impegnativo lavorare con i dati nella forma in cui sono forniti:
who
#> # A tibble: 7,240 × 60
#> country iso2 iso3 year new_sp…¹ new_s…² new_s…³ new_s…⁴ new_s…⁵ new_s…⁶
#> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 Afghanistan AF AFG 1980 NA NA NA NA NA NA
#> 2 Afghanistan AF AFG 1981 NA NA NA NA NA NA
#> 3 Afghanistan AF AFG 1982 NA NA NA NA NA NA
#> 4 Afghanistan AF AFG 1983 NA NA NA NA NA NA
#> 5 Afghanistan AF AFG 1984 NA NA NA NA NA NA
#> 6 Afghanistan AF AFG 1985 NA NA NA NA NA NA
#> # … with 7,234 more rows, 50 more variables: new_sp_m65 <dbl>,
#> # new_sp_f014 <dbl>, new_sp_f1524 <dbl>, new_sp_f2534 <dbl>,
#> # new_sp_f3544 <dbl>, new_sp_f4554 <dbl>, new_sp_f5564 <dbl>,
#> # new_sp_f65 <dbl>, new_sn_m014 <dbl>, new_sn_m1524 <dbl>,
#> # new_sn_m2534 <dbl>, new_sn_m3544 <dbl>, new_sn_m4554 <dbl>,
#> # new_sn_m5564 <dbl>, new_sn_m65 <dbl>, new_sn_f014 <dbl>,
#> # new_sn_f1524 <dbl>, new_sn_f2534 <dbl>, new_sn_f3544 <dbl>, …Questo è un tipico set di dati della vita reale. Contiene colonne ridondanti, codici variabili strani e molti valori mancanti. In breve, who è disordinato, e avremo bisogno di più passi per metterlo in ordine. Come dplyr, tidyr è progettato in modo che ogni funzione faccia bene una cosa. Questo significa che in situazioni reali avrete bisogno di mettere insieme più verbi in una pipeline.
Il posto migliore per iniziare è quasi sempre quello di riunire le colonne che non sono variabili. Diamo un’occhiata a quello che abbiamo:
Sembra che
country,iso2eiso3siano tre variabili che specificano in modo ridondante il paese.Anche
yearè chiaramente una variabile.Non sappiamo ancora quali siano tutte le altre colonne, ma data la struttura nei nomi delle variabili (es.
new_sp_m014,new_ep_m014,new_ep_f014) è probabile che questi siano valori, non variabili.
Quindi abbiamo bisogno di raccogliere tutte le colonne da new_sp_m014 a newrel_f65. Non sappiamo ancora cosa rappresentano questi valori, quindi daremo loro il nome generico di "key". Sappiamo che le celle rappresentano il numero di casi, quindi useremo la variabile cases. Ci sono molti valori mancanti nella rappresentazione attuale, quindi per ora useremo values_drop_na solo per concentrarci sui valori che sono presenti.
who1 <- who %>%
pivot_longer(
cols = new_sp_m014:newrel_f65,
names_to = "key",
values_to = "cases",
values_drop_na = TRUE
)
who1
#> # A tibble: 76,046 × 6
#> country iso2 iso3 year key cases
#> <chr> <chr> <chr> <dbl> <chr> <dbl>
#> 1 Afghanistan AF AFG 1997 new_sp_m014 0
#> 2 Afghanistan AF AFG 1997 new_sp_m1524 10
#> 3 Afghanistan AF AFG 1997 new_sp_m2534 6
#> 4 Afghanistan AF AFG 1997 new_sp_m3544 3
#> 5 Afghanistan AF AFG 1997 new_sp_m4554 5
#> 6 Afghanistan AF AFG 1997 new_sp_m5564 2
#> # … with 76,040 more rowsPossiamo avere qualche indizio sulla struttura dei valori nella nuova colonna “key” contandoli:
who1 %>%
count(key)
#> # A tibble: 56 × 2
#> key n
#> <chr> <int>
#> 1 new_ep_f014 1032
#> 2 new_ep_f1524 1021
#> 3 new_ep_f2534 1021
#> 4 new_ep_f3544 1021
#> 5 new_ep_f4554 1017
#> 6 new_ep_f5564 1017
#> # … with 50 more rowsPotresti essere in grado di analizzarlo da solo con un po’ di ragionamento e qualche esperimento, ma fortunatamente abbiamo il dizionario dei dati a portata di mano. Ci dice:
Le prime tre lettere di ogni colonna indicano se la colonna contiene nuovi o vecchi casi di TBC. In questo set di dati, ogni colonna contiene nuovi casi.
-
Le due lettere successive descrivono il tipo di TBC:
-
relsta per i casi di ricaduta -
epsta per i casi di TB extrapolmonare -
snsta per i casi di TB polmonare che non possono essere diagnosticati da uno striscio polmonare (striscio negativo) -
spsta per i casi di TB polmonare che potrebbero essere diagnosticati da uno striscio polmonare (striscio positivo)
-
La sesta lettera indica il sesso dei pazienti con TB. Il dataset raggruppa casi per maschi (
m) e femmine (f).-
I numeri rimanenti danno il gruppo di età. Il set di dati raggruppa i casi in sette gruppi di età:
-
014= 0 – 14 anni -
1524= 15 – 24 anni -
2534= 25 – 34 anni -
3544= 35 – 44 anni -
4554= 45 – 54 anni -
5564= 55 – 64 anni -
65= 65 anni o più
-
Dobbiamo fare una piccola correzione al formato dei nomi delle colonne: sfortunatamente i nomi sono leggermente incoerenti perché invece di new_rel abbiamo newrel (è difficile da notare qui, ma se non lo correggi avremo degli errori nei passi successivi). Imparerai a conoscere str_replace() in [strings], ma l’idea di base è abbastanza semplice: sostituisci i caratteri “newrel” con “new_rel”. Questo rende coerenti tutti i nomi delle variabili.
who2 <- who1 %>%
mutate(key = stringr::str_replace(key, "newrel", "new_rel"))
who2
#> # A tibble: 76,046 × 6
#> country iso2 iso3 year key cases
#> <chr> <chr> <chr> <dbl> <chr> <dbl>
#> 1 Afghanistan AF AFG 1997 new_sp_m014 0
#> 2 Afghanistan AF AFG 1997 new_sp_m1524 10
#> 3 Afghanistan AF AFG 1997 new_sp_m2534 6
#> 4 Afghanistan AF AFG 1997 new_sp_m3544 3
#> 5 Afghanistan AF AFG 1997 new_sp_m4554 5
#> 6 Afghanistan AF AFG 1997 new_sp_m5564 2
#> # … with 76,040 more rowsPossiamo separare i valori in ogni codice con due passaggi di separate(). Il primo passaggio dividerà i codici ad ogni trattino basso.
who3 <- who2 %>%
separate(key, c("new", "type", "sexage"), sep = "_")
who3
#> # A tibble: 76,046 × 8
#> country iso2 iso3 year new type sexage cases
#> <chr> <chr> <chr> <dbl> <chr> <chr> <chr> <dbl>
#> 1 Afghanistan AF AFG 1997 new sp m014 0
#> 2 Afghanistan AF AFG 1997 new sp m1524 10
#> 3 Afghanistan AF AFG 1997 new sp m2534 6
#> 4 Afghanistan AF AFG 1997 new sp m3544 3
#> 5 Afghanistan AF AFG 1997 new sp m4554 5
#> 6 Afghanistan AF AFG 1997 new sp m5564 2
#> # … with 76,040 more rowsAllora potremmo anche eliminare la colonna new perché è costante in questo set di dati. Mentre eliminiamo le colonne, eliminiamo anche iso2 e iso3 perché sono ridondanti.
who3 %>%
count(new)
#> # A tibble: 1 × 2
#> new n
#> <chr> <int>
#> 1 new 76046
who4 <- who3 %>%
select(-new, -iso2, -iso3)Poi separeremo sexage in sex e age dividendo dopo il primo carattere:
who5 <- who4 %>%
separate(sexage, c("sex", "age"), sep = 1)
who5
#> # A tibble: 76,046 × 6
#> country year type sex age cases
#> <chr> <dbl> <chr> <chr> <chr> <dbl>
#> 1 Afghanistan 1997 sp m 014 0
#> 2 Afghanistan 1997 sp m 1524 10
#> 3 Afghanistan 1997 sp m 2534 6
#> 4 Afghanistan 1997 sp m 3544 3
#> 5 Afghanistan 1997 sp m 4554 5
#> 6 Afghanistan 1997 sp m 5564 2
#> # … with 76,040 more rowsThe who dataset is now tidy!
Vi ho mostrato il codice un pezzo alla volta, assegnando ogni risultato intermedio ad una nuova variabile. Questo tipicamente non è il modo in cui si lavora in modo interattivo. Invece, costruireste gradualmente un tubo complesso:
who %>%
pivot_longer(
cols = new_sp_m014:newrel_f65,
names_to = "key",
values_to = "cases",
values_drop_na = TRUE
) %>%
mutate(
key = stringr::str_replace(key, "newrel", "new_rel")
) %>%
separate(key, c("new", "var", "sexage")) %>%
select(-new, -iso2, -iso3) %>%
separate(sexage, c("sex", "age"), sep = 1)12.6.1 Esercizi
In questo caso di studio ho impostato
values_drop_na = TRUEsolo per rendere più facile controllare che avessimo i valori corretti. È ragionevole? Pensate a come sono rappresentati i valori mancanti in questo set di dati. Ci sono valori impliciti valori mancanti? Qual è la differenza tra unNAe uno zero?Cosa succede se trascurate il passo
mutate()? (mutate(names_from = stringr::str_replace(key, "newrel", "new_rel")))Ho affermato che
iso2eiso3sono ridondanti concountry. Confermate questa affermazione.Per ogni paese, anno e sesso, calcola il numero totale di casi di TBC. Fai una visualizzazione informativa dei dati.
12.7 Dati non Tidy
Prima di continuare con altri argomenti, vale la pena di parlare brevemente dei dati non ordinati. All’inizio del capitolo, ho usato il termine peggiorativo “disordinato” per riferirmi ai dati non ordinati. Questa è una semplificazione eccessiva: ci sono molte strutture di dati utili e ben fondate che non sono dati ordinati. Ci sono due ragioni principali per usare altre strutture di dati:
Rappresentazioni alternative possono avere prestazioni sostanziali o vantaggi di spazio. vantaggi di spazio.
I campi specializzati hanno sviluppato le proprie convenzioni per la memorizzazione dei dati che possono essere molto diverse dalle convenzioni dei dati ordinati.
Ognuno di questi motivi significa che avrete bisogno di qualcosa di diverso da un tibble (o data frame). Se i vostri dati si adattano naturalmente ad una struttura rettangolare composta da osservazioni e variabili, penso che tidy data dovrebbe essere la vostra scelta predefinita. Ma ci sono buone ragioni per usare altre strutture; i dati ordinati non sono l’unico modo.
Se volete saperne di più sui dati non ordinati, vi consiglio vivamente questo post sul blog di Jeff Leek: http://simplystatistics.org/2016/02/17/non-tidy-data/.