21 Iterazioni
21.1 Introduzione
In funzioni, abbiamo parlato di quanto sia importante ridurre la duplicazione del codice creando funzioni invece di copiare e incollare. Ridurre la duplicazione del codice ha tre benefici principali:
È più facile vedere l’intento del vostro codice, perché i vostri occhi sono attratti da ciò che è diverso, non da ciò che rimane lo stesso.
È più facile rispondere ai cambiamenti dei requisiti. Quando i tuoi bisogni esigenze, hai solo bisogno di fare cambiamenti in un posto, piuttosto che ricordarsi di cambiare ogni posto in cui hai copiato e incollato il codice.
È probabile che abbiate meno bug perché ogni linea di codice è usata in più posti.
Uno strumento per ridurre la duplicazione sono le funzioni, che riducono la duplicazione identificando schemi ripetuti di codice e li estraggono in pezzi indipendenti che possono essere facilmente riutilizzati e aggiornati. Un altro strumento per ridurre la duplicazione è l’ iterazione, che vi aiuta quando avete bisogno di fare la stessa cosa a più input: ripetere la stessa operazione su diverse colonne, o su diversi insiemi di dati. In questo capitolo imparerete due importanti paradigmi di iterazione: la programmazione imperativa e la programmazione funzionale. Sul lato imperativo ci sono strumenti come i cicli for e i cicli while, che sono un ottimo punto di partenza perché rendono l’iterazione molto esplicita, quindi è ovvio cosa sta succedendo. Tuttavia, i cicli for sono abbastanza prolissi e richiedono un bel po’ di codice di supporto che viene duplicato per ogni ciclo for. La programmazione funzionale (FP) offre strumenti per estrarre questo codice duplicato, così ogni comune modello di ciclo for ha la sua propria funzione. Una volta che si padroneggia il vocabolario della FP, si possono risolvere molti problemi comuni di iterazione con meno codice, più facilità e meno errori.
21.2 Cicli for
Immaginiamo di avere questa semplice tibble:
Vogliamo calcolare la mediana di ogni colonna. Si potrebbe fare con il copia-e-incolla:
median(df$a)
#> [1] -0.2457625
median(df$b)
#> [1] -0.2873072
median(df$c)
#> [1] -0.05669771
median(df$d)
#> [1] 0.1442633Ma questo infrange la nostra regola empirica: mai copiare e incollare più di due volte. Invece, potremmo usare un ciclo for:
output <- vector("double", ncol(df)) # 1. output
for (i in seq_along(df)) { # 2. sequenza
output[[i]] <- median(df[[i]]) # 3. corpo
}
output
#> [1] -0.24576245 -0.28730721 -0.05669771 0.14426335Ogni ciclo for ha tre componenti:
-
Il output:
output <- vector("double", length(x)). Prima di iniziare il ciclo, dovete sempre allocare uno spazio sufficiente per l’output. Questo è molto importante per l’efficienza: se fate crescere il ciclo for ad ogni iterazione usandoc()(per esempio), il vostro ciclo for sarà molto lento.Un modo generale per creare un vettore vuoto di una data lunghezza è la funzione
vector()è la funzionevector(). Ha due argomenti: il tipo di vettore (“logico”, “intero”, “doppio”, “carattere”, ecc.) e la lunghezza del vettore. -
La sequenza:
i in seq_along(df). Questo determina su cosa eseguire il ciclo: ogni esecuzione del ciclo for assegnerà aiun valore diverso daseq_along(df). È utile pensare aicome a un pronome, come “it”.Potreste non aver visto
seq_along()prima. È una versione sicura del familiare1:length(l), con un’importante differenza: se avete un vettore di lunghezza zero,seq_along()fa la cosa giusta:Probabilmente non creerete deliberatamente un vettore di lunghezza zero, ma è facile crearli accidentalmente. Se usate
1:length(x)invece di diseq_along(x), probabilmente otterrete un messaggio di errore confuso. Il corpo:
output[[i]] <- mediana(df[[i]]). Questo è il codice che fa il lavoro. Viene eseguito ripetutamente, ogni volta con un valore diverso peri. La prima iterazione eseguiràoutput[[1]] <- mediana(df[[1]]), la seconda eseguiràoutput[[2]] <- mediana(df[[2]]), e così via.
Questo è tutto quello che c’è nel ciclo for! Ora è un buon momento per fare pratica nel creare alcuni cicli for di base (e non così di base) usando gli esercizi qui sotto. Poi passeremo ad alcune variazioni del ciclo for che vi aiuteranno a risolvere altri problemi che si presenteranno nella pratica.
21.2.1 Esercizi
-
Scrivere cicli for per:
- Calcolare la media di ogni colonna in
mtcars. - Determinare il tipo di ogni colonna in
nycflights13::flights. - Calcolare il numero di valori unici in ogni colonna di
iris. - Genera 10 normali casuali da distribuzioni con medie di -10, 0, 10 e 100.
Pensate all’output, alla sequenza e al corpo prima di iniziare a scrivere il ciclo.
- Calcolare la media di ogni colonna in
-
Eliminate il ciclo for in ognuno dei seguenti esempi sfruttando sfruttando una funzione esistente che lavora con i vettori:
out <- "" for (x in letters) { out <- stringr::str_c(out, x) } x <- sample(100) sd <- 0 for (i in seq_along(x)) { sd <- sd + (x[i] - mean(x)) ^ 2 } sd <- sqrt(sd / (length(x) - 1)) x <- runif(100) out <- vector("numeric", length(x)) out[1] <- x[1] for (i in 2:length(x)) { out[i] <- out[i - 1] + x[i] } -
Combinate le vostre abilità di scrittura di funzioni e di ciclo for:
Scrivi un ciclo for che
stampi()il testo della canzone per bambini “Alice il cammello”.Converti la filastrocca “dieci nel letto” in una funzione. Generalizzare a qualsiasi numero di persone in qualsiasi struttura per dormire.
Convertire la canzone “99 bottiglie di birra sul muro” in una funzione. Generalizzare a qualsiasi numero di qualsiasi recipiente contenente qualsiasi liquido su qualsiasi superficie.
-
È comune vedere cicli for che non preallocano l’output e invece aumentano la lunghezza di un vettore ad ogni passo:
output <- vector("integer", 0) for (i in seq_along(x)) { output <- c(output, lengths(x[[i]])) } outputCome influisce sulle prestazioni? Progettate ed eseguite un esperimento.
21.3 Variazioni del ciclo for
Una volta che avete il ciclo for di base sotto la vostra cintura, ci sono alcune variazioni di cui dovreste essere consapevoli. Queste variazioni sono importanti indipendentemente da come si fa l’iterazione, quindi non dimenticatele una volta che avete imparato le tecniche FP che imparerete nella prossima sezione.
Ci sono quattro variazioni sul tema di base del ciclo for:
- Modificare un oggetto esistente, invece di crearne uno nuovo.
- Looping su nomi o valori, invece che su indici.
- Gestire uscite di lunghezza sconosciuta.
- Gestione di sequenze di lunghezza sconosciuta.
21.3.1 Modifica di un oggetto esistente
A volte volete usare un ciclo for per modificare un oggetto esistente. Per esempio, ricordate la nostra sfida da funzioni. Volevamo ridimensionare ogni colonna di un frame di dati:
df <- tibble(
a = rnorm(10),
b = rnorm(10),
c = rnorm(10),
d = rnorm(10)
)
rescale01 <- function(x) {
rng <- range(x, na.rm = TRUE)
(x - rng[1]) / (rng[2] - rng[1])
}
df$a <- rescale01(df$a)
df$b <- rescale01(df$b)
df$c <- rescale01(df$c)
df$d <- rescale01(df$d)Per risolvere questo con un ciclo for pensiamo di nuovo ai tre componenti:
Output: abbiamo già l’output — è uguale all’input!
Sequenza: possiamo pensare a un data frame come a una lista di colonne, quindi possiamo iterare su ogni colonna con
seq_along(df).Corpo: applicare
rescale01().
Questo ci dà:
for (i in seq_along(df)) {
df[[i]] <- rescale01(df[[i]])
}Tipicamente modificherete una lista o un data frame con questo tipo di ciclo, quindi ricordatevi di usare [[, non [. Potreste aver notato che ho usato [[ in tutti i miei cicli for: Penso sia meglio usare [[ anche per i vettori atomici perché rende chiaro che voglio lavorare con un singolo elemento.
21.3.2 Modelli di looping
Ci sono tre modi di base per fare un loop su un vettore. Finora vi ho mostrato il più generale: il looping sugli indici numerici con for (i in seq_along(xs)), e l’estrazione del valore con x[[i]]. Ci sono altre due forme:
Loop sugli elementi:
for (x in xs). Questo è più utile se vi interessa solo preoccuparsi solo degli effetti collaterali, come tracciare o salvare un file, perché è difficile salvare l’output in modo efficiente.-
Fate un loop sui nomi:
for (nm in names(xs)). Questo vi dà il nome, che potete usare per accedere al valore conx[[nm]]. Questo è utile se volete usare il nome nel titolo di un grafico o nel nome di un file. Se state creando un output con nome, assicuratevi di nominare il vettore dei risultati in questo modo:
L’iterazione sugli indici numerici è la forma più generale, perché data la posizione si può estrarre sia il nome che il valore:
21.3.3 Lunghezza di uscita sconosciuta
A volte potreste non sapere quanto sarà lungo l’output. Per esempio, immaginate di voler simulare alcuni vettori casuali di lunghezza casuale. Potreste essere tentati di risolvere questo problema facendo crescere progressivamente il vettore:
means <- c(0, 1, 2)
output <- double()
for (i in seq_along(means)) {
n <- sample(100, 1)
output <- c(output, rnorm(n, means[[i]]))
}
str(output)
#> num [1:138] 0.912 0.205 2.584 -0.789 0.588 ...Ma questo non è molto efficiente perché in ogni iterazione, R deve copiare tutti i dati dalle iterazioni precedenti. In termini tecnici si ottiene un comportamento “quadratico” (\(O(n^2)\)) che significa che un ciclo con un numero di elementi tre volte superiore richiederebbe nove (\(3^2\)) volte più tempo per essere eseguito.
Una soluzione migliore è quella di salvare i risultati in una lista, e poi combinarli in un singolo vettore dopo che il ciclo è finito:
out <- vector("list", length(means))
for (i in seq_along(means)) {
n <- sample(100, 1)
out[[i]] <- rnorm(n, means[[i]])
}
str(out)
#> List of 3
#> $ : num [1:76] -0.3389 -0.0756 0.0402 0.1243 -0.9984 ...
#> $ : num [1:17] -0.11 1.149 0.614 0.77 1.392 ...
#> $ : num [1:41] 1.88 2.46 2.62 1.82 1.88 ...
str(unlist(out))
#> num [1:134] -0.3389 -0.0756 0.0402 0.1243 -0.9984 ...Qui ho usato unlist() per appiattire una lista di vettori in un singolo vettore. Un’opzione più rigorosa è quella di usare purrr::flatten_dbl() — esso darà un errore se l’input non è una lista di double.
Questo schema si verifica anche in altri posti:
Potreste generare una lunga stringa. Invece di
paste()insieme ogni iterazione con la precedente, salvate l’output in un vettore di caratteri e poi combinate quel vettore in una singola stringa conpaste(output, collapse = "").Potreste generare un grande frame di dati. Invece di sequenziare
rbind()in ogni iterazione, salvate l’output in una lista, poi usatedplyr::bind_rows(output)per combinare l’output in un singolo frame di dati.
Fate attenzione a questo schema. Ogni volta che lo vedete, passate ad un oggetto risultato più complesso, e poi combinate in un solo passo alla fine.
21.3.4 Lunghezza della sequenza sconosciuta
A volte non sapete nemmeno per quanto tempo la sequenza di input debba essere eseguita. Questo è comune quando si fanno simulazioni. Per esempio, potreste voler fare un ciclo finché non ottenete tre teste in fila. Non potete fare questo tipo di iterazione con il ciclo for. Invece, potete usare un ciclo while. Un ciclo while è più semplice del ciclo for perché ha solo due componenti, una condizione e un corpo:
while (condition) {
# corpo
}Un ciclo while è anche più generale di un ciclo for, perché potete riscrivere qualsiasi ciclo for come un ciclo while, ma non potete riscrivere ogni ciclo while come un ciclo for:
for (i in seq_along(x)) {
# corpo
}
# Equivalente a
i <- 1
while (i <= length(x)) {
# corpo
i <- i + 1
}Ecco come potremmo usare un ciclo while per trovare quanti tentativi ci vogliono per ottenere tre teste di fila:
flip <- function() sample(c("T", "H"), 1)
flips <- 0
nheads <- 0
while (nheads < 3) {
if (flip() == "H") {
nheads <- nheads + 1
} else {
nheads <- 0
}
flips <- flips + 1
}
flips
#> [1] 21Menziono i cicli while solo brevemente, perché non li uso quasi mai. Sono più spesso usati per la simulazione, che è al di fuori dello scopo di questo libro. Tuttavia, è bene sapere che esistono in modo da essere preparati per problemi in cui il numero di iterazioni non è noto in anticipo.
21.3.5 Esercizi
Immaginate di avere una directory piena di file CSV che volete leggere. Avete i loro percorsi in un vettore,
files <- dir("data/", pattern = "\\csv$", full.names = TRUE), e ora vuole leggere ognuno di essi conread_csv(). Scrivete il ciclo for che li carichi in un singolo frame di dati.Cosa succede se usate
for (nm in names(x))exnon ha nomi? Cosa succede se solo alcuni degli elementi hanno un nome? Cosa succede se i nomi non sono non sono unici?-
Scrivete una funzione che stampi la media di ogni colonna numerica in un data dati, insieme al suo nome. Per esempio,
show_mean(iris)stamperebbe:show_mean(iris) #> Sepal.Length: 5.84 #> Sepal.Width: 3.06 #> Petal.Length: 3.76 #> Petal.Width: 1.20(Sfida extra: quale funzione ho usato per assicurarmi che i numeri fossero ben allineati, anche se i nomi delle variabili avevano lunghezze diverse?)
-
Cosa fa questo codice? Come funziona?
21.4 Cicli for vs. funzionali
I cicli for non sono così importanti in R come lo sono in altri linguaggi perché R è un linguaggio di programmazione funzionale. Questo significa che è possibile avvolgere i cicli for in una funzione e chiamare quella funzione invece di usare direttamente il ciclo for.
Per vedere perché questo è importante, considerate (di nuovo) questo semplice frame di dati:
Immaginate di voler calcolare la media di ogni colonna. Potreste farlo con un ciclo for:
output <- vector("double", length(df))
for (i in seq_along(df)) {
output[[i]] <- mean(df[[i]])
}
output
#> [1] -0.3260369 0.1356639 0.4291403 -0.2498034Vi rendete conto che vorrete calcolare la media di ogni colonna abbastanza frequentemente, quindi la estraete in una funzione:
col_mean <- function(df) {
output <- vector("double", length(df))
for (i in seq_along(df)) {
output[i] <- mean(df[[i]])
}
output
}Ma poi pensate che sarebbe utile poter calcolare anche la mediana e la deviazione standard, quindi copiate e incollate la vostra funzione col_mean() e sostituite la mean() con median() e sd():
col_median <- function(df) {
output <- vector("double", length(df))
for (i in seq_along(df)) {
output[i] <- median(df[[i]])
}
output
}
col_sd <- function(df) {
output <- vector("double", length(df))
for (i in seq_along(df)) {
output[i] <- sd(df[[i]])
}
output
}Uh oh! Avete copiato e incollato questo codice due volte, quindi è il momento di pensare a come generalizzarlo. Notate che la maggior parte di questo codice è un boilerplate for-loop ed è difficile vedere l’unica cosa (mean(), median(), sd()) che è diversa tra le funzioni.
Cosa fareste se vedeste una serie di funzioni come questa:
f1 <- function(x) abs(x - mean(x)) ^ 1
f2 <- function(x) abs(x - mean(x)) ^ 2
f3 <- function(x) abs(x - mean(x)) ^ 3Si spera di notare che ci sono molti doppioni e di estrarli in un argomento aggiuntivo:
Avete ridotto la possibilità di generare bug (perché ora avete 1/3 del codice originale), e lo avete reso facile da generalizzare a nuove situazioni.
Possiamo fare esattamente la stessa cosa con col_mean(), col_median() e col_sd() aggiungendo un argomento che fornisce la funzione da applicare ad ogni colonna:
col_summary <- function(df, fun) {
out <- vector("double", length(df))
for (i in seq_along(df)) {
out[i] <- fun(df[[i]])
}
out
}
col_summary(df, median)
#> [1] -0.51850298 0.02779864 0.17295591 -0.61163819
col_summary(df, mean)
#> [1] -0.3260369 0.1356639 0.4291403 -0.2498034L’idea di passare una funzione ad un’altra funzione è un’idea estremamente potente, ed è uno dei comportamenti che rende R un linguaggio di programmazione funzionale. Potrebbe volerci un po’ di tempo per comprendere l’idea, ma ne vale la pena. Nel resto del capitolo, conoscerete e userete il pacchetto purrr, che fornisce funzioni che eliminano la necessità di molti comuni cicli for. La famiglia di funzioni apply in R base (apply(), lapply(), tapply(), etc) risolve un problema simile, ma purrr è più coerente e quindi più facile da imparare.
L’obiettivo di usare le funzioni purrr al posto dei cicli for è di permettervi di rompere le comuni sfide di manipolazione delle liste in pezzi indipendenti:
Come puoi risolvere il problema per un singolo elemento della lista? Una volta una volta risolto il problema, purrr si occupa di generalizzare la soluzione ad ogni elemento della lista.
Se stai risolvendo un problema complesso, come puoi scomporlo in pezzi che ti permettono di avanzare di un piccolo passo verso la soluzione? soluzione? Con Purrr, ottieni tanti piccoli pezzi che puoi comporre insieme con il tubo.
Questa struttura rende più facile risolvere nuovi problemi. Rende anche più facile capire le tue soluzioni ai vecchi problemi quando rileggi il tuo vecchio codice.
21.4.1 Esercizi
Leggete la documentazione per
apply(). Nel caso 2d, quali due cicli for generalizza?Adattate
col_summary()in modo che si applichi solo alle colonne numeriche Potreste voler iniziare con una funzioneis_numeric()che restituisca un vettore logico che ha un TRUE corrispondente ad ogni colonna numerica.
21.5 Le funzioni map
Lo schema di eseguire un ciclo su un vettore, fare qualcosa ad ogni elemento e salvare i risultati è così comune che il pacchetto purrr fornisce una famiglia di funzioni che lo fanno per voi. C’è una funzione per ogni tipo di output:
-
map()fa una lista. -
map_lgl()crea un vettore logico. -
map_int()crea un vettore intero. -
map_dbl()crea un vettore doppio. - La funzione
map_chr()crea un vettore di caratteri.
Ogni funzione prende un vettore come input, applica una funzione ad ogni pezzo, e poi restituisce un nuovo vettore che ha la stessa lunghezza (e gli stessi nomi) dell’input. Il tipo di vettore è determinato dal suffisso della funzione map.
Una volta che padroneggiate queste funzioni, scoprirete che ci vuole molto meno tempo per risolvere i problemi di iterazione. Ma non dovreste mai sentirvi in colpa se usate un ciclo for invece di una funzione map. Le funzioni mappa sono un passo avanti nella torre di astrazione, e può volerci molto tempo per capire come funzionano. L’importante è che risolviate il problema su cui state lavorando, non che scriviate il codice più conciso ed elegante (anche se è sicuramente qualcosa a cui volete tendere!).
Alcune persone vi diranno di evitare i cicli for perché sono lenti. Si sbagliano! (Beh, almeno sono piuttosto fuori moda, dato che i cicli for non sono stati lenti per molti anni). Il principale vantaggio di usare funzioni come map() non è la velocità, ma la chiarezza: rendono il vostro codice più facile da scrivere e da leggere.
Possiamo usare queste funzioni per eseguire gli stessi calcoli dell’ultimo ciclo for. Quelle funzioni di riepilogo hanno restituito dei doppi, quindi abbiamo bisogno di usare map_dbl():
map_dbl(df, mean)
#> a b c d
#> -0.3260369 0.1356639 0.4291403 -0.2498034
map_dbl(df, median)
#> a b c d
#> -0.51850298 0.02779864 0.17295591 -0.61163819
map_dbl(df, sd)
#> a b c d
#> 0.9214834 0.4848945 0.9816016 1.1563324Rispetto all’uso di un ciclo for, l’attenzione si concentra sull’operazione che viene eseguita (cioè mean(), median(), sd()), non sulla contabilità richiesta per fare il loop su ogni elemento e memorizzare l’output. Questo è ancora più evidente se usiamo la pipe:
df %>% map_dbl(mean)
#> a b c d
#> -0.3260369 0.1356639 0.4291403 -0.2498034
df %>% map_dbl(median)
#> a b c d
#> -0.51850298 0.02779864 0.17295591 -0.61163819
df %>% map_dbl(sd)
#> a b c d
#> 0.9214834 0.4848945 0.9816016 1.1563324Ci sono alcune differenze tra map_*() e col_summary():
Tutte le funzioni purrr sono implementate in C. Questo le rende un po’ più veloci a spese della leggibilità.
Il secondo argomento,
.f, la funzione da applicare, può essere una formula, un vettore di caratteri o un vettore di interi. Imparerete queste comode scorciatoie nella prossima sezione.-
map_*()usa … ([dot dot dot]) per passare ulteriori argomenti a.fogni volta che viene chiamato:map_dbl(df, mean, trim = 0.5) #> a b c d #> -0.51850298 0.02779864 0.17295591 -0.61163819 -
Le funzioni map conservano anche i nomi:
21.5.1 Scorciatoie
Ci sono alcune scorciatoie che potete usare con .f per risparmiare un po’ di battitura. Immaginate di voler adattare un modello lineare ad ogni gruppo in un set di dati. Il seguente esempio giocattolo divide il dataset mtcars in tre pezzi (uno per ogni valore di cilindro) e adatta lo stesso modello lineare ad ogni pezzo:
La sintassi per creare una funzione anonima in R è piuttosto prolissa, così purrr fornisce una comoda scorciatoia: una formula unilaterale.
Qui ho usato . come pronome: si riferisce all’elemento corrente della lista (nello stesso modo in cui i si riferisce all’indice corrente nel ciclo for).
Quando state guardando molti modelli, potreste voler estrarre una statistica riassuntiva come il \(R^2\). Per farlo dobbiamo prima eseguire summary() e poi estrarre il componente chiamato r.quadrato. Potremmo farlo usando la scorciatoia per le funzioni anonime:
Ma estrarre componenti con nome è un’operazione comune, quindi purrr fornisce una scorciatoia ancora più breve: si può usare una stringa.
Potete anche usare un intero per selezionare gli elementi in base alla posizione:
21.5.2 Base R
Se avete familiarità con la famiglia di funzioni apply in R base, potreste aver notato alcune somiglianze con le funzioni purrr:
lapply()è fondamentalmente identica amap(), eccetto chemap()è coerente con tutte le altre funzioni in purrr, e potete usare le scorciatoie per.f.-
Base
sapply()è un wrapper intorno alapply()che automaticamente semplifica l’output. Questo è utile per il lavoro interattivo ma è problematico in una funzione perché non si sa mai che tipo di output otterrete:x1 <- list( c(0.27, 0.37, 0.57, 0.91, 0.20), c(0.90, 0.94, 0.66, 0.63, 0.06), c(0.21, 0.18, 0.69, 0.38, 0.77) ) x2 <- list( c(0.50, 0.72, 0.99, 0.38, 0.78), c(0.93, 0.21, 0.65, 0.13, 0.27), c(0.39, 0.01, 0.38, 0.87, 0.34) ) threshold <- function(x, cutoff = 0.8) x[x > cutoff] x1 %>% sapply(threshold) %>% str() #> List of 3 #> $ : num 0.91 #> $ : num [1:2] 0.9 0.94 #> $ : num(0) x2 %>% sapply(threshold) %>% str() #> num [1:3] 0.99 0.93 0.87 vapply()è un’alternativa sicura asapply()perché si fornisce un argomento aggiuntivo che definisce il tipo. L’unico problema convapply()è che richiede un sacco di battitura:vapply(df, is.numeric, logical(1))è equivalente amap_lgl(df, is.numeric). Un vantaggio divapply()rispetto alle funzioni map è che può produrre anche matrici — le funzioni map producono solo producono solo vettori.
Mi concentro qui sulle funzioni di Purrr perché hanno nomi e argomenti più coerenti, utili scorciatoie, e in futuro forniranno un facile parallelismo e barre di progresso.
21.5.3 Esercizi
-
Scrivete del codice che usi una delle funzioni map per:
- Calcolare la media di ogni colonna in
mtcars.
- Calcolare la media di ogni colonna in
- Determinare il tipo di ogni colonna in
nycflights13::flights. - Calcolare il numero di valori unici in ogni colonna di
iris. - Genera 10 normali casuali da distribuzioni con medie di -10, 0, 10 e 100.
Come si può creare un singolo vettore che per ogni colonna di un data frame indichi se è un fattore o meno?
Cosa succede quando usi le funzioni map su vettori che non sono liste? Cosa fa la funzione
map(1:5, runif)? Perché?-
Cosa fa
map(-2:2, rnorm, n = 5)? Perché?- Che cosa fa
map_dbl(-2:2, rnorm, n = 5)? Perché?
- Che cosa fa
Riscrivete
map(x, function(df) lm(mpg ~ wt, data = df))per eliminare la funzione anonima.
21.6 Affrontare il fallimento
Quando usate le funzioni map per ripetere molte operazioni, le possibilità che una di queste operazioni fallisca sono molto più alte. Quando ciò accade, si ottiene un messaggio di errore e nessun output. Questo è fastidioso: perché un fallimento vi impedisce di accedere a tutti gli altri successi? Come ci si assicura che una mela marcia non rovini l’intero barile?
In questa sezione imparerete come affrontare questa situazione con una nuova funzione: safely(). La funzione safely() è un avverbio: prende una funzione (un verbo) e ne restituisce una versione modificata. In questo caso, la funzione modificata non darà mai un errore. Invece, restituisce sempre una lista con due elementi:
resultè il risultato originale. Se c’è stato un errore, questo saràNULL.errorè un oggetto di errore. Se l’operazione ha avuto successo, questo saràNULL.
(Potreste avere familiarità con la funzione try() in R base. È simile, ma poiché a volte restituisce il risultato originale e a volte restituisce un oggetto errore, è più difficile lavorarci).
Illustriamo questo con un semplice esempio: log():
safe_log <- safely(log)
str(safe_log(10))
#> List of 2
#> $ result: num 2.3
#> $ error : NULL
str(safe_log("a"))
#> List of 2
#> $ result: NULL
#> $ error :List of 2
#> ..$ message: chr "non-numeric argument to mathematical function"
#> ..$ call : language .Primitive("log")(x, base)
#> ..- attr(*, "class")= chr [1:3] "simpleError" "error" "condition"Quando la funzione ha successo, l’elemento result contiene il risultato e l’elemento error è NULL. Quando la funzione fallisce, l’elemento result è NULL e l’elemento error contiene un oggetto di errore.
La funzione safely() è progettata per lavorare con map:
x <- list(1, 10, "a")
y <- x %>% map(safely(log))
str(y)
#> List of 3
#> $ :List of 2
#> ..$ result: num 0
#> ..$ error : NULL
#> $ :List of 2
#> ..$ result: num 2.3
#> ..$ error : NULL
#> $ :List of 2
#> ..$ result: NULL
#> ..$ error :List of 2
#> .. ..$ message: chr "non-numeric argument to mathematical function"
#> .. ..$ call : language .Primitive("log")(x, base)
#> .. ..- attr(*, "class")= chr [1:3] "simpleError" "error" "condition"Sarebbe più facile lavorare con questo se avessimo due liste: una di tutti gli errori e una di tutti gli output. Questo è facile da ottenere con purrr::transpose():
y <- y %>% transpose()
str(y)
#> List of 2
#> $ result:List of 3
#> ..$ : num 0
#> ..$ : num 2.3
#> ..$ : NULL
#> $ error :List of 3
#> ..$ : NULL
#> ..$ : NULL
#> ..$ :List of 2
#> .. ..$ message: chr "non-numeric argument to mathematical function"
#> .. ..$ call : language .Primitive("log")(x, base)
#> .. ..- attr(*, "class")= chr [1:3] "simpleError" "error" "condition"Sta a te decidere come trattare gli errori, ma tipicamente guarderai i valori di x dove y è un errore, o lavorerai con i valori di y che sono ok:
is_ok <- y$error %>% map_lgl(is_null)
x[!is_ok]
#> [[1]]
#> [1] "a"
y$result[is_ok] %>% flatten_dbl()
#> [1] 0.000000 2.302585Purrr fornisce altri due utili avverbi:
-
Come
safely(),possibly()ha sempre successo. È più semplice disafely(), perché gli si dà un valore predefinito da restituire in caso di errore. -
quietly()svolge un ruolo simile a quello disafely(), ma invece di catturare gli errori, cattura l’output stampato, i messaggi e gli avvertimenti:
21.7 Map su più argomenti
Finora abbiamo mappato lungo un singolo input. Ma spesso avete più input correlati che avete bisogno di iterare in parallelo. Questo è il lavoro delle funzioni map2() e pmap(). Per esempio, immaginate di voler simulare alcune normali casuali con mezzi diversi. Sapete come farlo con map():
mu <- list(5, 10, -3)
mu %>%
map(rnorm, n = 5) %>%
str()
#> List of 3
#> $ : num [1:5] 5.63 7.1 4.39 3.37 4.99
#> $ : num [1:5] 9.34 9.33 9.52 11.32 10.64
#> $ : num [1:5] -2.49 -4.75 -2.11 -2.78 -2.42E se si volesse variare anche la deviazione standard? Un modo per farlo sarebbe quello di iterare sugli indici e indicizzare in vettori di medie e deviazioni standard:
sigma <- list(1, 5, 10)
seq_along(mu) %>%
map(~rnorm(5, mu[[.]], sigma[[.]])) %>%
str()
#> List of 3
#> $ : num [1:5] 4.82 5.74 4 2.06 5.72
#> $ : num [1:5] 6.51 0.529 10.381 14.377 12.269
#> $ : num [1:5] -11.51 2.66 8.52 -10.56 -7.89Ma questo offusca l’intento del codice. Invece potremmo usare map2() che itera su due vettori in parallelo:
map2(mu, sigma, rnorm, n = 5) %>% str()
#> List of 3
#> $ : num [1:5] 3.83 4.52 5.12 3.23 3.59
#> $ : num [1:5] 13.55 3.8 8.16 12.31 8.39
#> $ : num [1:5] -15.872 -13.3 12.141 0.469 14.794map2() genera questa serie di chiamate di funzioni:
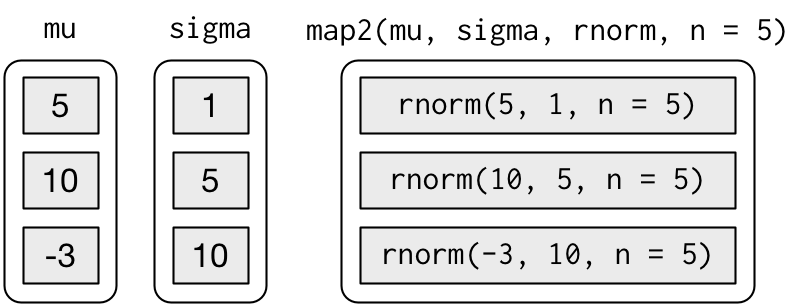
Notate che gli argomenti che variano per ogni chiamata vengono prima della funzione; gli argomenti che sono gli stessi per ogni chiamata vengono dopo.
Come map(), map2() è solo un involucro attorno ad un ciclo for:
map2 <- function(x, y, f, ...) {
out <- vector("list", length(x))
for (i in seq_along(x)) {
out[[i]] <- f(x[[i]], y[[i]], ...)
}
out
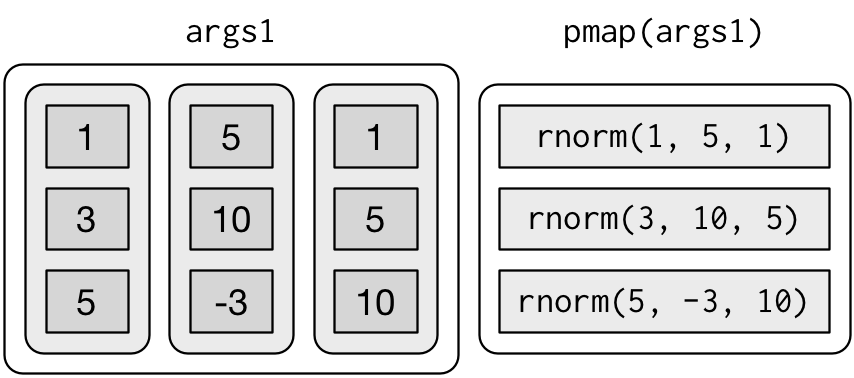
}Si potrebbe anche immaginare map3(), map4(), map5(), map6() ecc. Invece, purrr fornisce pmap() che prende una lista di argomenti. Potreste usarlo se voleste variare la media, la deviazione standard e il numero di campioni:
n <- list(1, 3, 5)
args1 <- list(n, mu, sigma)
args1 %>%
pmap(rnorm) %>%
str()
#> List of 3
#> $ : num 5.39
#> $ : num [1:3] 5.41 2.08 9.58
#> $ : num [1:5] -23.85 -2.96 -6.56 8.46 -5.21Questo sembra:
Se non nominate gli elementi della lista, pmap() userà la corrispondenza posizionale quando chiama la funzione. Questo è un po’ fragile e rende il codice più difficile da leggere, quindi è meglio nominare gli argomenti:
Questo genera chiamate più lunghe, ma più sicure:
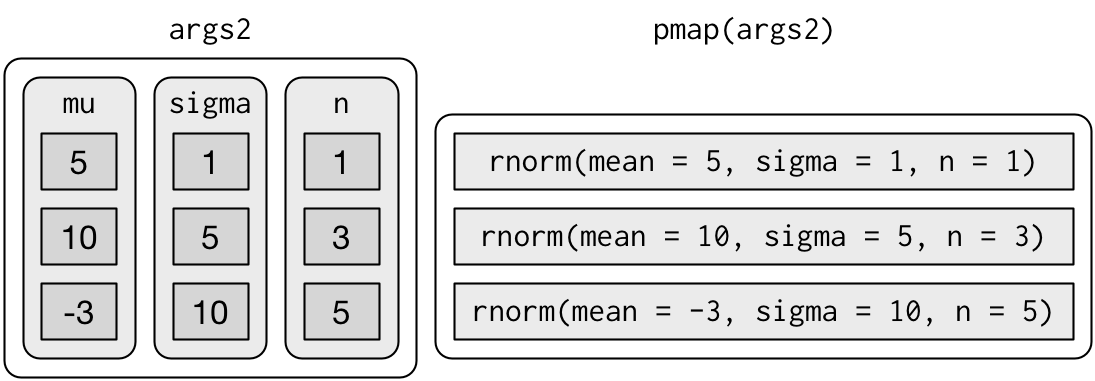
Poiché gli argomenti sono tutti della stessa lunghezza, ha senso memorizzarli in un frame di dati:
params <- tribble(
~mean, ~sd, ~n,
5, 1, 1,
10, 5, 3,
-3, 10, 5
)
params %>%
pmap(rnorm)
#> [[1]]
#> [1] 6.018179
#>
#> [[2]]
#> [1] 8.681404 18.292712 6.129566
#>
#> [[3]]
#> [1] -12.239379 -5.755334 -8.933997 -4.222859 8.797842Non appena il vostro codice diventa complicato, penso che un data frame sia un buon approccio perché assicura che ogni colonna abbia un nome e sia della stessa lunghezza di tutte le altre colonne.
21.7.1 Invocare diverse funzioni
C’è un ulteriore passo avanti nella complessità: oltre a variare gli argomenti della funzione, potreste anche variare la funzione stessa:
f <- c("runif", "rnorm", "rpois")
param <- list(
list(min = -1, max = 1),
list(sd = 5),
list(lambda = 10)
)Per gestire questo caso, potete usare invoke_map():
invoke_map(f, param, n = 5) %>% str()
#> Warning: `invoke_map()` was deprecated in purrr 1.0.0.
#> ℹ Please use map() + exec() instead.
#> List of 3
#> $ : num [1:5] 0.479 0.439 -0.471 0.348 -0.581
#> $ : num [1:5] 2.48 3.9 7.54 -9.12 3.94
#> $ : int [1:5] 6 11 5 8 9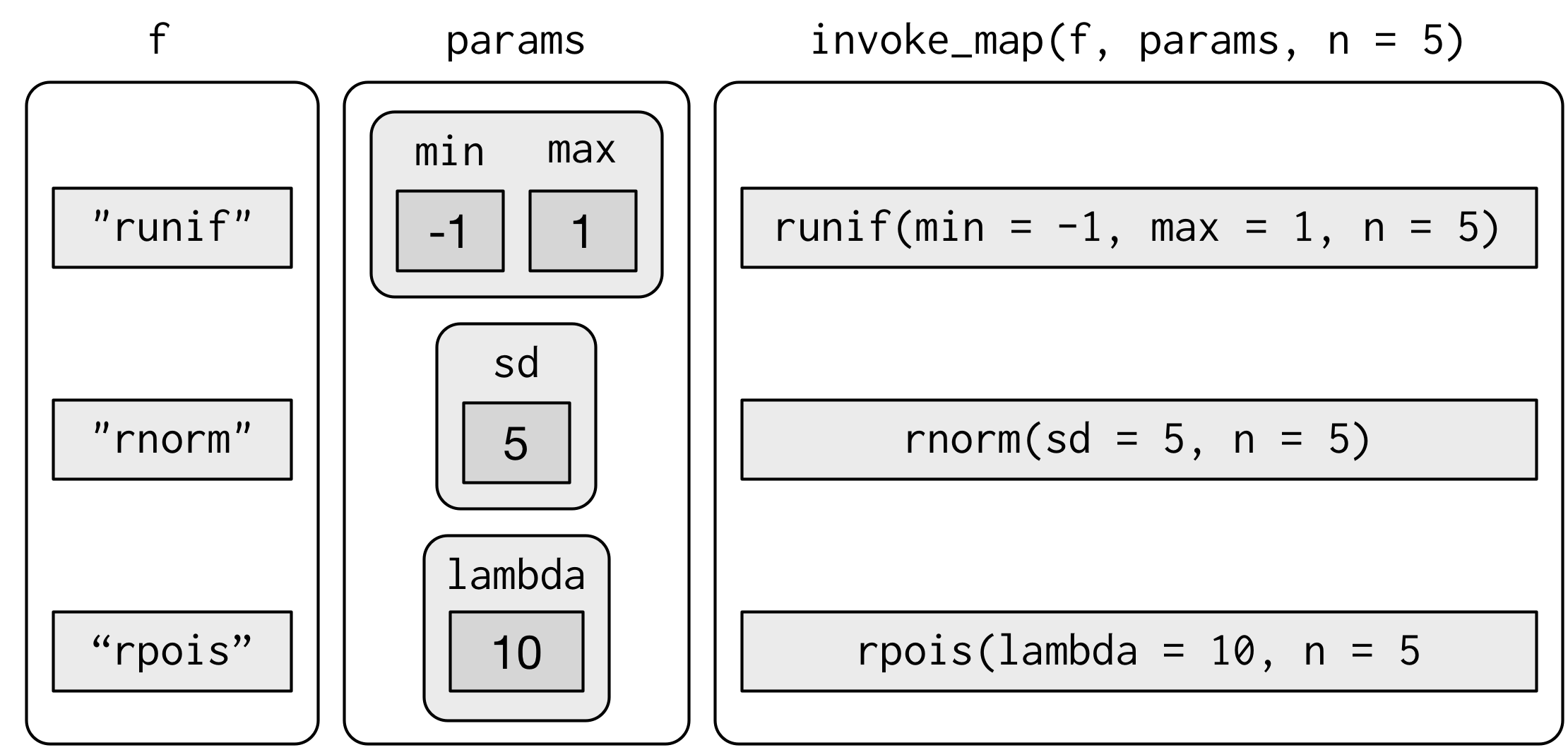
Il primo argomento è una lista di funzioni o un vettore di caratteri di nomi di funzioni. Il secondo argomento è una lista di liste che danno gli argomenti che variano per ogni funzione. Gli argomenti successivi sono passati ad ogni funzione.
E ancora, potete usare tribble() per rendere la creazione di queste coppie di argomenti un po’ più facile:
21.8 Walk
Walk è un’alternativa a map che si usa quando si vuole chiamare una funzione per i suoi effetti collaterali, piuttosto che per il suo valore di ritorno. Di solito lo fai perché vuoi rendere l’output sullo schermo o salvare file su disco - la cosa importante è l’azione, non il valore di ritorno. Ecco un esempio molto semplice:
walk() non è generalmente molto utile rispetto a walk2() o pwalk(). Per esempio, se avete una lista di trame e un vettore di nomi di file, potreste usare pwalk() per salvare ogni file nella posizione corrispondente sul disco:
library(ggplot2)
plots <- mtcars %>%
split(.$cyl) %>%
map(~ggplot(., aes(mpg, wt)) + geom_point())
paths <- stringr::str_c(names(plots), ".pdf")
pwalk(list(paths, plots), ggsave, path = tempdir())walk(), walk2() e pwalk() restituiscono tutti invisibilmente .x, il primo argomento. Questo li rende adatti all’uso nel mezzo delle pipeline.
21.9 Altri modelli di cicli for
Purrr fornisce un certo numero di altre funzioni che astraggono da altri tipi di cicli for. Le userai meno frequentemente delle funzioni di mappa, ma sono utili da conoscere. L’obiettivo qui è di illustrare brevemente ogni funzione, così speriamo che vi venga in mente se vedete un problema simile in futuro. Poi si può andare a cercare la documentazione per maggiori dettagli.
21.9.1 Funzioni predicate
Un certo numero di funzioni lavorano con funzioni predicate che restituiscono un singolo TRUE o FALSE.
Le funzioni keep() e discard() mantengono gli elementi dell’input dove il predicato è rispettivamente TRUE o FALSE:
iris %>%
keep(is.factor) %>%
str()
#> 'data.frame': 150 obs. of 1 variable:
#> $ Species: Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 1 1 1 ...
iris %>%
discard(is.factor) %>%
str()
#> 'data.frame': 150 obs. of 4 variables:
#> $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ...
#> $ Sepal.Width : num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ...
#> $ Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ...
#> $ Petal.Width : num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ...some() e every() determinano se il predicato è vero per qualsiasi o per tutti gli elementi.
x <- list(1:5, letters, list(10))
x %>%
some(is_character)
#> [1] TRUE
x %>%
every(is_vector)
#> [1] TRUEdetect() trova il primo elemento in cui il predicato è vero; detect_index() restituisce la sua posizione.
x <- sample(10)
x
#> [1] 10 6 1 3 2 4 5 8 9 7
x %>%
detect(~ . > 5)
#> [1] 10
x %>%
detect_index(~ . > 5)
#> [1] 1head_while() e tail_while() prendono elementi dall’inizio o dalla fine di un vettore mentre un predicato è vero:
x %>%
head_while(~ . > 5)
#> [1] 10 6
x %>%
tail_while(~ . > 5)
#> [1] 8 9 721.9.2 Ridurre e accumulare
A volte si ha una lista complessa che si vuole ridurre ad una lista semplice applicando ripetutamente una funzione che riduce una coppia ad un singoletto. Questo è utile se si vuole applicare un verbo di dplyr a due tabelle a più tabelle. Per esempio, potreste avere una lista di frame di dati, e volete ridurla ad un singolo frame di dati unendo gli elementi insieme:
dfs <- list(
age = tibble(name = "John", age = 30),
sex = tibble(name = c("John", "Mary"), sex = c("M", "F")),
trt = tibble(name = "Mary", treatment = "A")
)
dfs %>% reduce(full_join)
#> Joining with `by = join_by(name)`
#> Joining with `by = join_by(name)`
#> # A tibble: 2 × 4
#> name age sex treatment
#> <chr> <dbl> <chr> <chr>
#> 1 John 30 M <NA>
#> 2 Mary NA F AO forse avete una lista di vettori e volete trovare l’intersezione:
vs <- list(
c(1, 3, 5, 6, 10),
c(1, 2, 3, 7, 8, 10),
c(1, 2, 3, 4, 8, 9, 10)
)
vs %>% reduce(intersect)
#> [1] 1 3 10La funzione reduce() prende una funzione “binaria” (cioè una funzione con due input primari), e la applica ripetutamente ad una lista finché non rimane un solo elemento.
La funzione accumulate() è simile, ma mantiene tutti i risultati intermedi. Potreste usarla per implementare una somma cumulativa:
x <- sample(10)
x
#> [1] 7 5 10 9 8 3 1 4 2 6
x %>% accumulate(`+`)
#> [1] 7 12 22 31 39 42 43 47 49 5521.9.3 Esercizi
Implementate la vostra versione di
every()usando un ciclo for. Confrontatela conpurrr::every(). Cosa fa la versione di Purrr che la vostra non fa?Create un
col_summary()migliorato che applica una funzione di riepilogo ad ogni colonna numerica in un frame di dati.-
Un possibile equivalente in R di base di
col_summary()è:col_sum3 <- function(df, f) { is_num <- sapply(df, is.numeric) df_num <- df[, is_num] sapply(df_num, f) }Ma ha una serie di bug come illustrato con i seguenti input:
df <- tibble( x = 1:3, y = 3:1, z = c("a", "b", "c") ) # OK col_sum3(df, mean) # Ha problemi: non sempre restituisce un vettore numerico col_sum3(df[1:2], mean) col_sum3(df[1], mean) col_sum3(df[0], mean)Cosa causa i bug?