25 Molti modelli
25.1 Introduzione
In questo capitolo imparerete tre potenti idee che vi aiuteranno a lavorare con un gran numero di modelli con facilità:
Usare molti modelli semplici per capire meglio insiemi di dati complessi.
Usare liste-colonne per memorizzare strutture di dati arbitrarie in un frame di dati. Per esempio, questo vi permetterà di avere una colonna che contiene modelli modelli lineari.
Usare il pacchetto broom, di David Robinson, per trasformare i modelli in dati dati ordinati. Questa è una tecnica potente per lavorare con un gran numero di modelli perché una volta che avete dati ordinati, potete applicare tutte le tecniche che avete imparato in precedenza nel libro.
Inizieremo con un esempio motivante utilizzando i dati sull’aspettativa di vita nel mondo. Si tratta di un piccolo set di dati, ma illustra quanto sia importante la modellazione per migliorare le vostre visualizzazioni. Useremo un gran numero di modelli semplici per separare alcuni dei segnali più forti in modo da poter vedere i segnali più sottili che rimangono. Vedremo anche come le sintesi dei modelli possono aiutarci a individuare i valori anomali e le tendenze insolite.
Le sezioni seguenti si immergeranno in maggiori dettagli sulle singole tecniche:
In list-columns, imparerai di più sulla struttura dati list-columns (‘colonne-elenco’), e perché ha senso mettere le liste nei frame di dati.
In [creare list-columns], imparerai i tre modi principali in cui creare liste-colonne.
In semplificare le list-columns imparerai come convertire le list-columns in vettori atomici regolari (o insiemi di vettori atomici) in modo da poter lavorare con loro più facilmente.
In rendere ordinati i dati con broom, imparerai a conoscere il set completo di strumenti forniti da broom e vedrai come possono essere applicati ad altri tipi di strutture dati.
Questo capitolo è in qualche modo aspirazionale: se questo libro è la vostra prima introduzione a R, questo capitolo sarà probabilmente una lotta. Richiede che abbiate idee profondamente interiorizzate su modellazione, strutture di dati e iterazione. Quindi non preoccupatevi se non lo capite — mettete da parte questo capitolo per qualche mese, e tornate quando vorrete ampliare il vostro cervello.
25.2 gapminder
Per motivare la potenza di molti modelli semplici, esamineremo i dati “gapminder”. Questi dati sono stati resi popolari da Hans Rosling, un medico e statistico svedese. Se non avete mai sentito parlare di lui, smettete subito di leggere questo capitolo e andate a guardare uno dei suoi video! È un fantastico presentatore di dati e illustra come è possibile utilizzare i dati per presentare una storia convincente. Un buon punto di partenza è questo breve video girato insieme alla BBC: https://www.youtube.com/watch?v=jbkSRLYSojo.
I dati di gapminder riassumono la progressione dei paesi nel tempo, guardando statistiche come l’aspettativa di vita e il PIL. I dati sono facilmente accessibili in R, grazie a Jenny Bryan che ha creato il pacchetto gapminder:
library(gapminder)
gapminder
#> # A tibble: 1,704 × 6
#> country continent year lifeExp pop gdpPercap
#> <fct> <fct> <int> <dbl> <int> <dbl>
#> 1 Afghanistan Asia 1952 28.8 8425333 779.
#> 2 Afghanistan Asia 1957 30.3 9240934 821.
#> 3 Afghanistan Asia 1962 32.0 10267083 853.
#> 4 Afghanistan Asia 1967 34.0 11537966 836.
#> 5 Afghanistan Asia 1972 36.1 13079460 740.
#> 6 Afghanistan Asia 1977 38.4 14880372 786.
#> # … with 1,698 more rowsIn questo caso di studio, ci concentreremo solo su tre variabili per rispondere alla domanda “Come cambia l’aspettativa di vita (lifeExp) nel tempo (year) per ogni paese (country)? Un buon punto di partenza è un grafico:
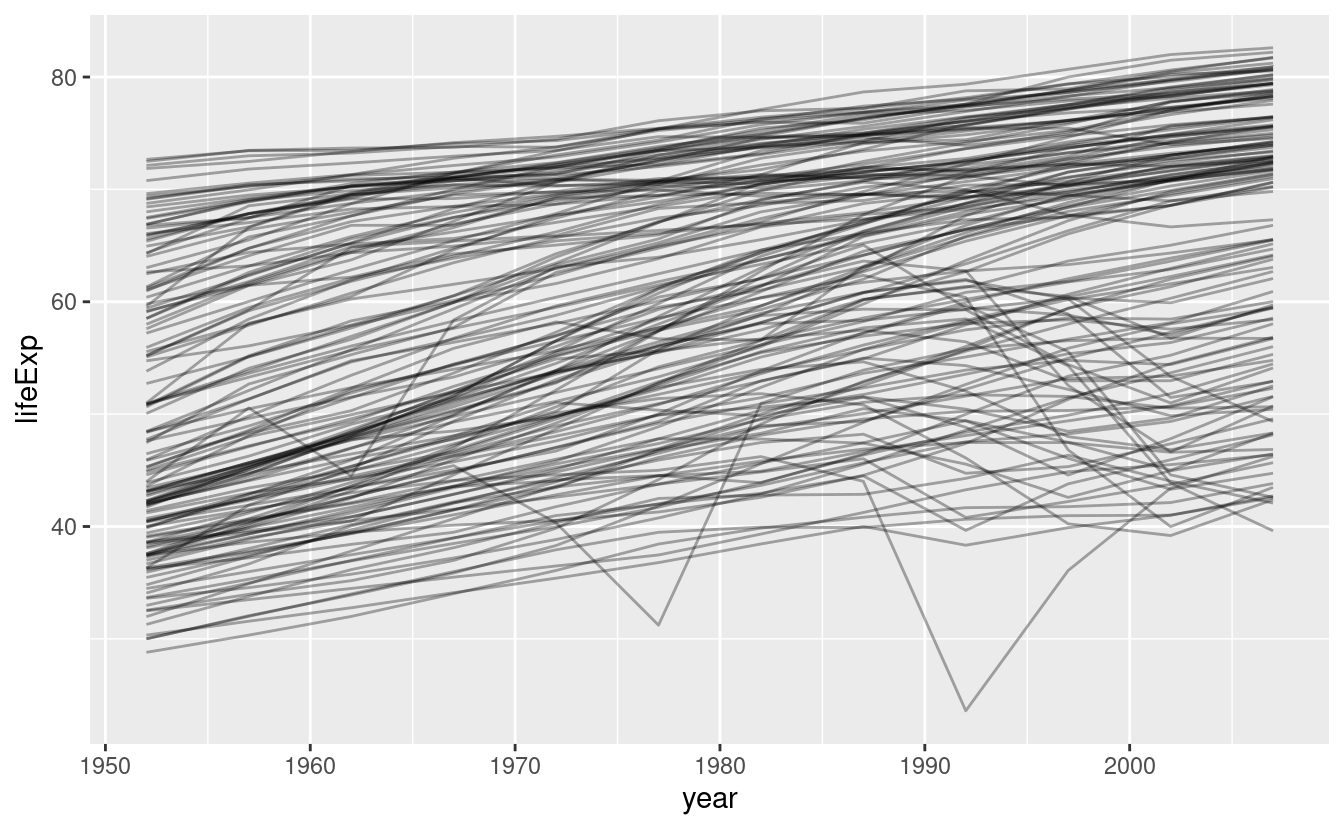
Questo è un piccolo dataset: ha solo ~1.700 osservazioni e 3 variabili. Ma è ancora difficile vedere cosa sta succedendo! Nel complesso, sembra che l’aspettativa di vita sia migliorata costantemente. Tuttavia, se si guarda da vicino, si possono notare alcuni paesi che non seguono questo schema. Come possiamo rendere questi paesi più facili da vedere?
Un modo è usare lo stesso approccio dell’ultimo capitolo: c’è un segnale forte (la crescita lineare complessiva) che rende difficile vedere tendenze più sottili. Distingueremo questi fattori adattando un modello con una tendenza lineare. Il modello cattura la crescita costante nel tempo, e i residui mostreranno ciò che rimane.
Sapete già come farlo se avessimo un singolo paese:
nz <- filter(gapminder, country == "New Zealand")
nz %>%
ggplot(aes(year, lifeExp)) +
geom_line() +
ggtitle("Full data = ")
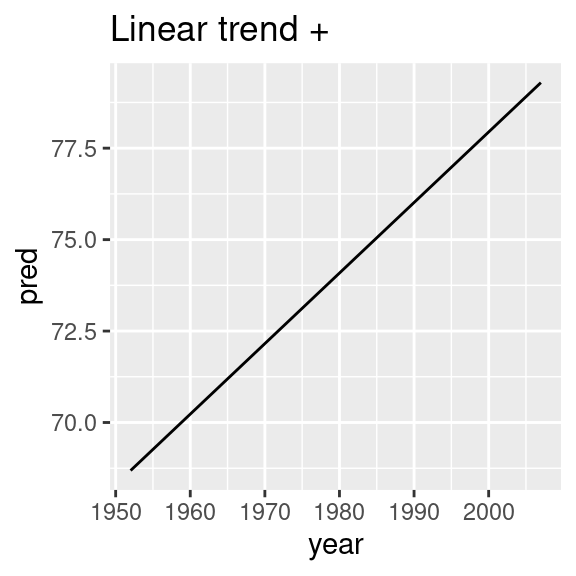
nz_mod <- lm(lifeExp ~ year, data = nz)
nz %>%
add_predictions(nz_mod) %>%
ggplot(aes(year, pred)) +
geom_line() +
ggtitle("Linear trend + ")
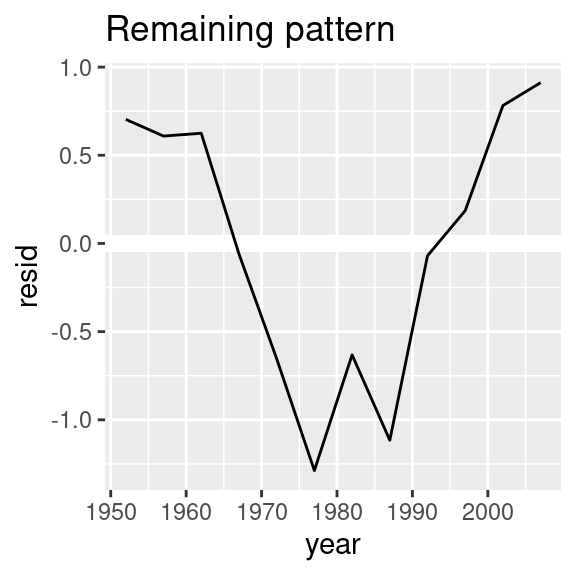
nz %>%
add_residuals(nz_mod) %>%
ggplot(aes(year, resid)) +
geom_hline(yintercept = 0, colour = "white", size = 3) +
geom_line() +
ggtitle("Remaining pattern")
#> Warning: Using `size` aesthetic for lines was deprecated in ggplot2 3.4.0.
#> ℹ Please use `linewidth` instead.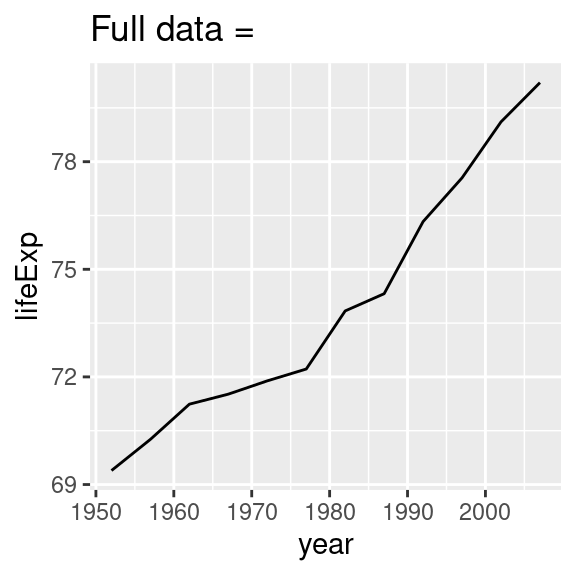
Come possiamo facilmente adattare questo modello ad ogni paese?
25.2.1 Dati annidati
Potresti immaginare di copiare e incollare quel codice più volte; ma hai già imparato un modo migliore! Estrai il codice comune con una funzione e ripeti usando una funzione di mappa da purrr. Questo problema è strutturato un po’ diversamente da quello che hai visto prima. Invece di ripetere un’azione per ogni variabile, vogliamo ripetere un’azione per ogni paese, un sottoinsieme di righe. Per farlo, abbiamo bisogno di una nuova struttura di dati: il data frame annidato (nested data frame). Per creare un data frame annidato, iniziamo con un data frame raggruppato e lo annidiamo:
by_country <- gapminder %>%
group_by(country, continent) %>%
nest()
by_country
#> # A tibble: 142 × 3
#> # Groups: country, continent [142]
#> country continent data
#> <fct> <fct> <list>
#> 1 Afghanistan Asia <tibble [12 × 4]>
#> 2 Albania Europe <tibble [12 × 4]>
#> 3 Algeria Africa <tibble [12 × 4]>
#> 4 Angola Africa <tibble [12 × 4]>
#> 5 Argentina Americas <tibble [12 × 4]>
#> 6 Australia Oceania <tibble [12 × 4]>
#> # … with 136 more rows(Sto imbrogliando un po’ raggruppando sia il continent che il country. Dato il country, il continente è fisso, quindi questo non aggiunge altri gruppi, ma è un modo semplice per portare con sé una variabile extra).
Questo crea un frame di dati che ha una riga per gruppo (per paese) e una colonna piuttosto insolita: data. data è una lista di data frame (o tibbie, per essere precisi). Sembra un’idea folle: abbiamo un data frame con una colonna che è una lista di altri data frame! Spiegherò brevemente perché penso che sia una buona idea.
La colonna data è un po’ difficile da guardare perché è una lista moderatamente complicata, e stiamo ancora lavorando su buoni strumenti per esplorare questi oggetti. Sfortunatamente l’uso di str() non è raccomandato perché spesso produce un output molto lungo. Ma se si estrae un singolo elemento dalla colonna data si vedrà che contiene tutti i dati per quel paese (in questo caso, Afghanistan).
by_country$data[[1]]
#> # A tibble: 12 × 4
#> year lifeExp pop gdpPercap
#> <int> <dbl> <int> <dbl>
#> 1 1952 28.8 8425333 779.
#> 2 1957 30.3 9240934 821.
#> 3 1962 32.0 10267083 853.
#> 4 1967 34.0 11537966 836.
#> 5 1972 36.1 13079460 740.
#> 6 1977 38.4 14880372 786.
#> # … with 6 more rowsNotate la differenza tra un data frame standard raggruppato e un data frame annidato: in un data frame raggruppato, ogni riga è un’osservazione; in un data frame annidato, ogni riga è un gruppo. Un altro modo di pensare a un dataset annidato è che ora abbiamo una meta-osservazione: una riga che rappresenta l’intero corso del tempo per un paese, piuttosto che un singolo punto nel tempo.
25.2.2 List-columns
Ora che abbiamo il nostro dataset annidato, siamo in una buona posizione per adattare alcuni modelli. Abbiamo una funzione di adattamento del modello:
country_model <- function(df) {
lm(lifeExp ~ year, data = df)
}E vogliamo applicarlo ad ogni frame di dati. I frame di dati sono in una lista, quindi possiamo usare purrr::map() per applicare country_model ad ogni elemento:
models <- map(by_country$data, country_model)Tuttavia, piuttosto che lasciare l’elenco dei modelli come un oggetto fluttuante, penso che sia meglio memorizzarlo come una colonna nel frame di dati by_country. Memorizzare oggetti correlati in colonne è una parte fondamentale del valore dei data frame, e il motivo per cui penso che le colonne-elenco siano una buona idea. Nel corso del lavoro con questi paesi, avremo molte liste dove abbiamo un elemento per paese. Quindi perché non memorizzarli tutti insieme in un unico frame di dati?
In altre parole, invece di creare un nuovo oggetto nell’ambiente globale, creeremo una nuova variabile nel data frame by_country. Questo è un lavoro per dplyr::mutate():
by_country <- by_country %>%
mutate(model = map(data, country_model))
by_country
#> # A tibble: 142 × 4
#> # Groups: country, continent [142]
#> country continent data model
#> <fct> <fct> <list> <list>
#> 1 Afghanistan Asia <tibble [12 × 4]> <lm>
#> 2 Albania Europe <tibble [12 × 4]> <lm>
#> 3 Algeria Africa <tibble [12 × 4]> <lm>
#> 4 Angola Africa <tibble [12 × 4]> <lm>
#> 5 Argentina Americas <tibble [12 × 4]> <lm>
#> 6 Australia Oceania <tibble [12 × 4]> <lm>
#> # … with 136 more rowsQuesto ha un grande vantaggio: poiché tutti gli oggetti correlati sono memorizzati insieme, non è necessario mantenerli manualmente sincronizzati quando si filtra o si organizza. La semantica del data frame se ne occupa per voi:
by_country %>%
filter(continent == "Europe")
#> # A tibble: 30 × 4
#> # Groups: country, continent [30]
#> country continent data model
#> <fct> <fct> <list> <list>
#> 1 Albania Europe <tibble [12 × 4]> <lm>
#> 2 Austria Europe <tibble [12 × 4]> <lm>
#> 3 Belgium Europe <tibble [12 × 4]> <lm>
#> 4 Bosnia and Herzegovina Europe <tibble [12 × 4]> <lm>
#> 5 Bulgaria Europe <tibble [12 × 4]> <lm>
#> 6 Croatia Europe <tibble [12 × 4]> <lm>
#> # … with 24 more rows
by_country %>%
arrange(continent, country)
#> # A tibble: 142 × 4
#> # Groups: country, continent [142]
#> country continent data model
#> <fct> <fct> <list> <list>
#> 1 Algeria Africa <tibble [12 × 4]> <lm>
#> 2 Angola Africa <tibble [12 × 4]> <lm>
#> 3 Benin Africa <tibble [12 × 4]> <lm>
#> 4 Botswana Africa <tibble [12 × 4]> <lm>
#> 5 Burkina Faso Africa <tibble [12 × 4]> <lm>
#> 6 Burundi Africa <tibble [12 × 4]> <lm>
#> # … with 136 more rowsSe la vostra lista di data frame e la lista di modelli fossero oggetti separati, dovete ricordarvi che ogni volta che riordinate o sotto-ordinate un vettore, dovete ri-ordinare o sotto-ordinare tutti gli altri per tenerli sincronizzati. Se lo dimenticate, il vostro codice continuerà a funzionare, ma darà la risposta sbagliata!
25.2.3 Disannidare (unnesting)
In precedenza abbiamo calcolato i residui di un singolo modello con un singolo set di dati. Ora abbiamo 142 frame di dati e 142 modelli. Per calcolare i residui, dobbiamo chiamare add_residuals() con ogni coppia modello-dati:
by_country <- by_country %>%
mutate(
resids = map2(data, model, add_residuals)
)
by_country
#> # A tibble: 142 × 5
#> # Groups: country, continent [142]
#> country continent data model resids
#> <fct> <fct> <list> <list> <list>
#> 1 Afghanistan Asia <tibble [12 × 4]> <lm> <tibble [12 × 5]>
#> 2 Albania Europe <tibble [12 × 4]> <lm> <tibble [12 × 5]>
#> 3 Algeria Africa <tibble [12 × 4]> <lm> <tibble [12 × 5]>
#> 4 Angola Africa <tibble [12 × 4]> <lm> <tibble [12 × 5]>
#> 5 Argentina Americas <tibble [12 × 4]> <lm> <tibble [12 × 5]>
#> 6 Australia Oceania <tibble [12 × 4]> <lm> <tibble [12 × 5]>
#> # … with 136 more rowsMa come si può tracciare una lista di frame di dati? Invece di lottare per rispondere a questa domanda, trasformiamo di nuovo la lista di data frame in un normale data frame. In precedenza abbiamo usato nest() per trasformare un data frame regolare in un data frame annidato, e ora facciamo il contrario con unnest():
resids <- unnest(by_country, resids)
resids
#> # A tibble: 1,704 × 9
#> # Groups: country, continent [142]
#> country continent data model year lifeExp pop gdpPercap resid
#> <fct> <fct> <list> <list> <int> <dbl> <int> <dbl> <dbl>
#> 1 Afghanistan Asia <tibble> <lm> 1952 28.8 8425333 779. -1.11
#> 2 Afghanistan Asia <tibble> <lm> 1957 30.3 9240934 821. -0.952
#> 3 Afghanistan Asia <tibble> <lm> 1962 32.0 10267083 853. -0.664
#> 4 Afghanistan Asia <tibble> <lm> 1967 34.0 11537966 836. -0.0172
#> 5 Afghanistan Asia <tibble> <lm> 1972 36.1 13079460 740. 0.674
#> 6 Afghanistan Asia <tibble> <lm> 1977 38.4 14880372 786. 1.65
#> # … with 1,698 more rowsSi noti che ogni colonna regolare è ripetuta una volta per ogni riga della tibla annidata.
Ora che abbiamo una struttura di dati regolare, possiamo tracciare i residui:
resids %>%
ggplot(aes(year, resid)) +
geom_line(aes(group = country), alpha = 1 / 3) +
geom_smooth(se = FALSE)
#> `geom_smooth()` using method = 'gam' and formula = 'y ~ s(x, bs = "cs")'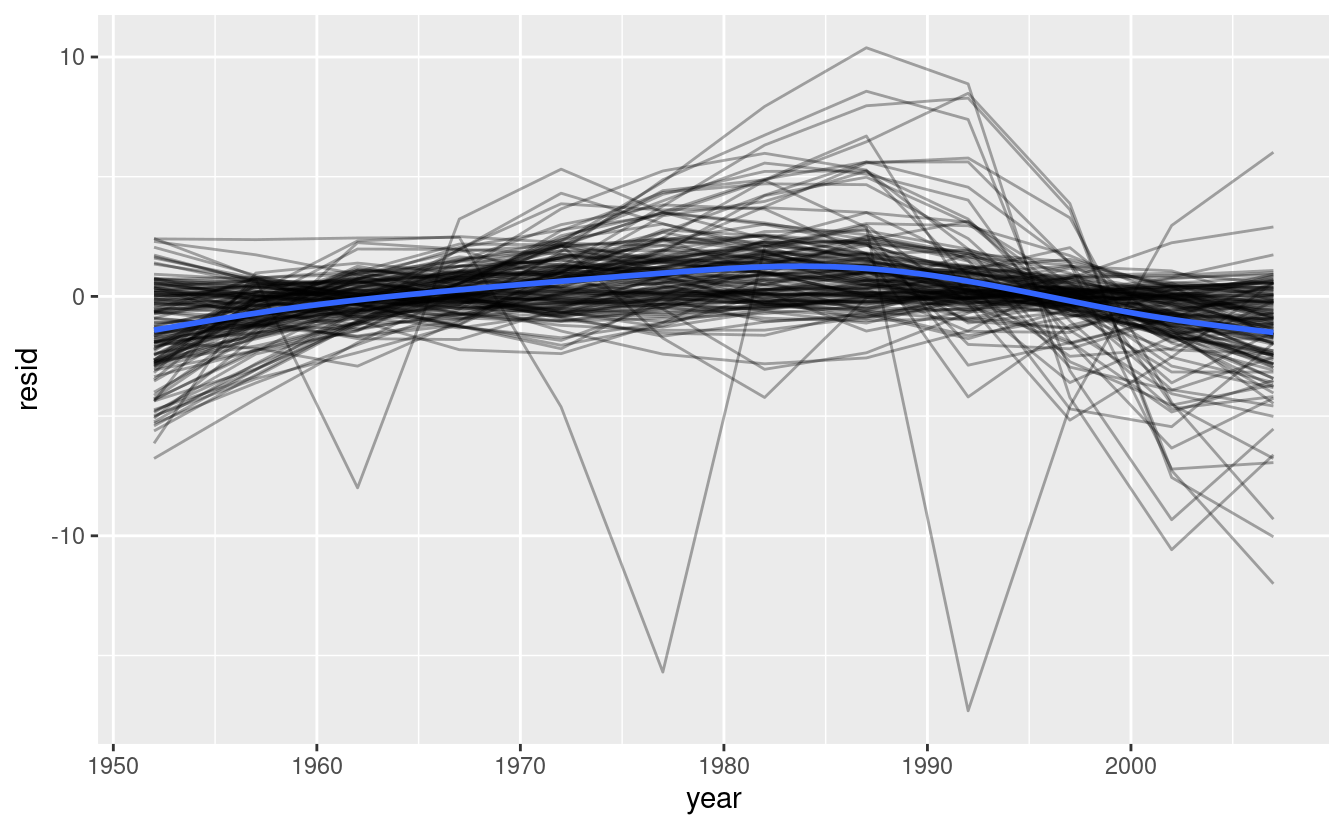
La sfaccettatura per continente è particolarmente rivelatrice:
resids %>%
ggplot(aes(year, resid, group = country)) +
geom_line(alpha = 1 / 3) +
facet_wrap(~continent)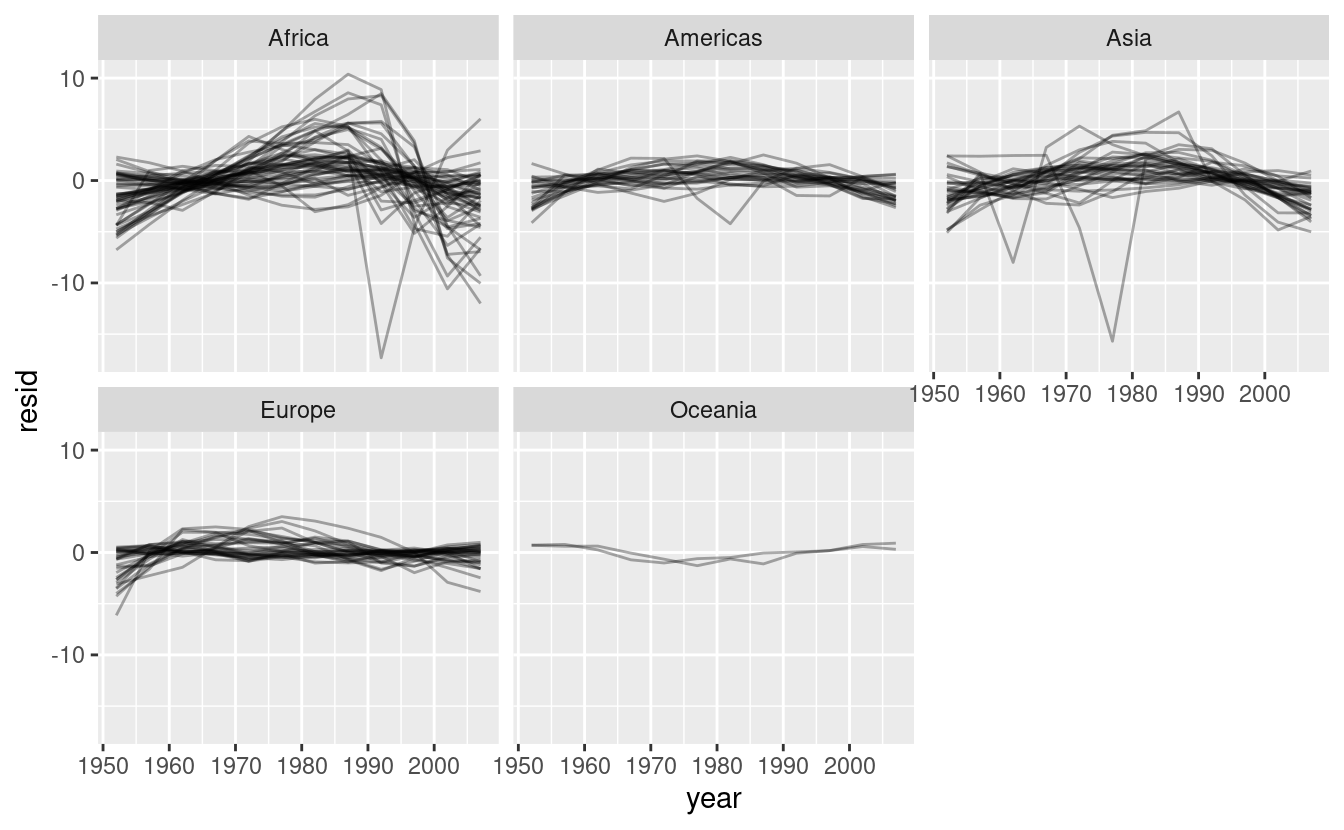
Sembra che ci siamo persi alcuni schemi leggeri. C’è anche qualcosa di interessante in Africa: vediamo alcuni residui molto grandi che suggeriscono che il nostro modello non si adatta così bene lì. Esploreremo meglio questo aspetto nella prossima sezione, attaccandolo da un’angolazione leggermente diversa.
25.2.4 Qualità del modello
Invece di guardare i residui del modello, potremmo guardare alcune misure generali della qualità del modello. Avete imparato come calcolare alcune misure specifiche nel capitolo precedente. Qui mostreremo un approccio diverso usando il pacchetto broom. Il pacchetto broom fornisce un insieme generale di funzioni per trasformare i modelli in dati ordinati. Qui useremo broom::glance() per estrarre alcune metriche di qualità del modello. Se lo applichiamo a un modello, otteniamo un frame di dati con una sola riga:
broom::glance(nz_mod)
#> # A tibble: 1 × 12
#> r.squ…¹ adj.r…² sigma stati…³ p.value df logLik AIC BIC devia…⁴ df.re…⁵
#> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int>
#> 1 0.954 0.949 0.804 205. 5.41e-8 1 -13.3 32.6 34.1 6.47 10
#> # … with 1 more variable: nobs <int>, and abbreviated variable names
#> # ¹r.squared, ²adj.r.squared, ³statistic, ⁴deviance, ⁵df.residualPossiamo usare mutate() e unnest() per creare un frame di dati con una riga per ogni paese:
by_country %>%
mutate(glance = map(model, broom::glance)) %>%
unnest(glance)
#> # A tibble: 142 × 17
#> # Groups: country, continent [142]
#> country conti…¹ data model resids r.squ…² adj.r…³ sigma stati…⁴ p.value
#> <fct> <fct> <list> <lis> <list> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 Afghan… Asia <tibble> <lm> <tibble> 0.948 0.942 1.22 181. 9.84e- 8
#> 2 Albania Europe <tibble> <lm> <tibble> 0.911 0.902 1.98 102. 1.46e- 6
#> 3 Algeria Africa <tibble> <lm> <tibble> 0.985 0.984 1.32 662. 1.81e-10
#> 4 Angola Africa <tibble> <lm> <tibble> 0.888 0.877 1.41 79.1 4.59e- 6
#> 5 Argent… Americ… <tibble> <lm> <tibble> 0.996 0.995 0.292 2246. 4.22e-13
#> 6 Austra… Oceania <tibble> <lm> <tibble> 0.980 0.978 0.621 481. 8.67e-10
#> # … with 136 more rows, 7 more variables: df <dbl>, logLik <dbl>, AIC <dbl>,
#> # BIC <dbl>, deviance <dbl>, df.residual <int>, nobs <int>, and abbreviated
#> # variable names ¹continent, ²r.squared, ³adj.r.squared, ⁴statisticQuesto non è esattamente l’output che vogliamo, perché include ancora tutte le colonne della lista. Questo è il comportamento predefinito quando unnest() lavora su frame di dati a riga singola. Per sopprimere queste colonne usiamo .drop = TRUE:
glance <- by_country %>%
mutate(glance = map(model, broom::glance)) %>%
unnest(glance, .drop = TRUE)
#> Warning: The `.drop` argument of `unnest()` is deprecated as of tidyr 1.0.0.
#> ℹ All list-columns are now preserved.
glance
#> # A tibble: 142 × 17
#> # Groups: country, continent [142]
#> country conti…¹ data model resids r.squ…² adj.r…³ sigma stati…⁴ p.value
#> <fct> <fct> <list> <lis> <list> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 Afghan… Asia <tibble> <lm> <tibble> 0.948 0.942 1.22 181. 9.84e- 8
#> 2 Albania Europe <tibble> <lm> <tibble> 0.911 0.902 1.98 102. 1.46e- 6
#> 3 Algeria Africa <tibble> <lm> <tibble> 0.985 0.984 1.32 662. 1.81e-10
#> 4 Angola Africa <tibble> <lm> <tibble> 0.888 0.877 1.41 79.1 4.59e- 6
#> 5 Argent… Americ… <tibble> <lm> <tibble> 0.996 0.995 0.292 2246. 4.22e-13
#> 6 Austra… Oceania <tibble> <lm> <tibble> 0.980 0.978 0.621 481. 8.67e-10
#> # … with 136 more rows, 7 more variables: df <dbl>, logLik <dbl>, AIC <dbl>,
#> # BIC <dbl>, deviance <dbl>, df.residual <int>, nobs <int>, and abbreviated
#> # variable names ¹continent, ²r.squared, ³adj.r.squared, ⁴statistic(Fate attenzione alle variabili che non sono stampate: c’è un sacco di roba utile lì).
Con questo quadro di dati in mano, possiamo iniziare a cercare i modelli che non si adattano bene:
glance %>%
arrange(r.squared)
#> # A tibble: 142 × 17
#> # Groups: country, continent [142]
#> country conti…¹ data model resids r.squ…² adj.r.…³ sigma stati…⁴ p.value
#> <fct> <fct> <list> <lis> <list> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 Rwanda Africa <tibble> <lm> <tibble> 0.0172 -0.0811 6.56 0.175 0.685
#> 2 Botswa… Africa <tibble> <lm> <tibble> 0.0340 -0.0626 6.11 0.352 0.566
#> 3 Zimbab… Africa <tibble> <lm> <tibble> 0.0562 -0.0381 7.21 0.596 0.458
#> 4 Zambia Africa <tibble> <lm> <tibble> 0.0598 -0.0342 4.53 0.636 0.444
#> 5 Swazil… Africa <tibble> <lm> <tibble> 0.0682 -0.0250 6.64 0.732 0.412
#> 6 Lesotho Africa <tibble> <lm> <tibble> 0.0849 -0.00666 5.93 0.927 0.358
#> # … with 136 more rows, 7 more variables: df <dbl>, logLik <dbl>, AIC <dbl>,
#> # BIC <dbl>, deviance <dbl>, df.residual <int>, nobs <int>, and abbreviated
#> # variable names ¹continent, ²r.squared, ³adj.r.squared, ⁴statisticI modelli peggiori sembrano essere tutti in Africa. Ricontrolliamo questo con un grafico. Qui abbiamo un numero relativamente piccolo di osservazioni e una variabile discreta, quindi geom_jitter() è efficace:
glance %>%
ggplot(aes(continent, r.squared)) +
geom_jitter(width = 0.5)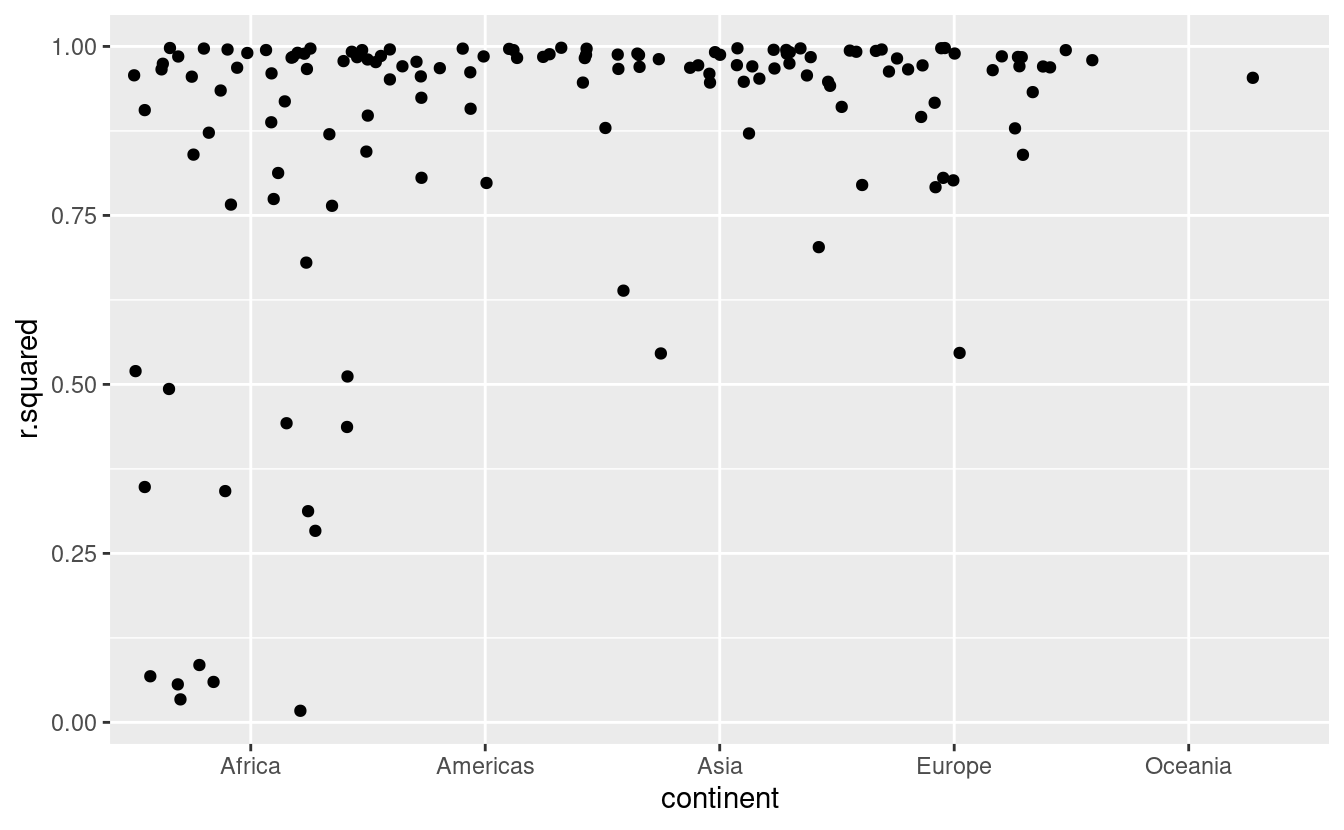
Potremmo estrarre i paesi con \(R^2\) particolarmente cattivi e tracciare i dati:
bad_fit <- filter(glance, r.squared < 0.25)
gapminder %>%
semi_join(bad_fit, by = "country") %>%
ggplot(aes(year, lifeExp, colour = country)) +
geom_line()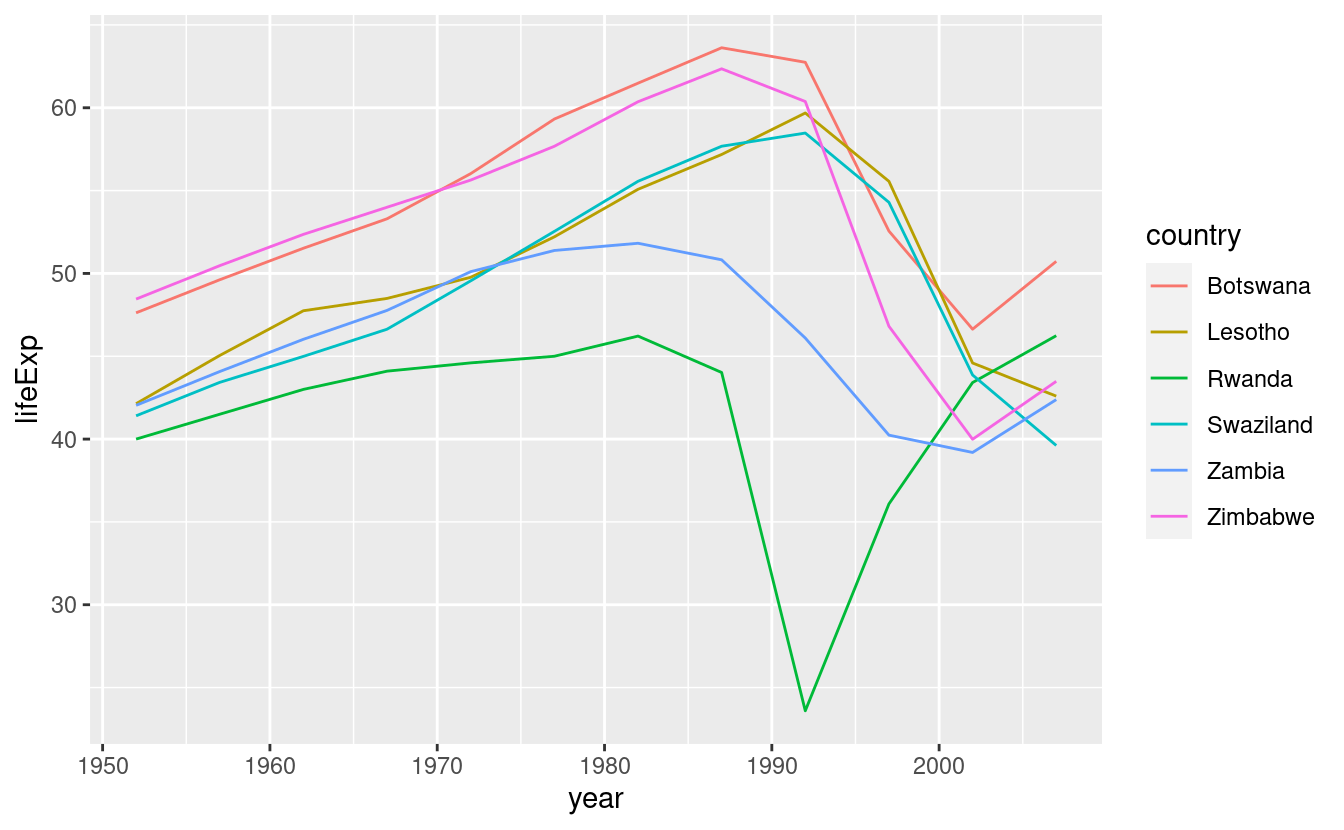
Qui vediamo due effetti principali: le tragedie dell’epidemia di HIV/AIDS e il genocidio del Ruanda.
25.2.5 Esercizi
Una tendenza lineare sembra essere un po’ troppo semplice per la tendenza generale. Puoi fare meglio con un polinomio quadratico? Come potete interpretare i coefficienti della quadratica? (Suggerimento: potreste voler trasformare anno` in modo che abbia media zero).
Esplorate altri metodi per visualizzare la distribuzione di \(R^2\) per continente. Potreste provare il pacchetto ggbeeswarm, che fornisce metodi simili per evitare sovrapposizioni come il jitter, ma usa metodi deterministici deterministici.
Per creare l’ultimo grafico (che mostra i dati per i paesi con il peggiori modelli di adattamento), abbiamo avuto bisogno di due passaggi: abbiamo creato un frame di dati con una riga per paese e poi lo abbiamo semi-unito al dataset originale. È possibile evitare questa unione se usiamo
unnest()invece diunnest(.drop = TRUE). Come?
25.3 List-columns
Ora che avete visto un flusso di lavoro di base per la gestione di molti modelli, rituffiamoci in alcuni dettagli. In questa sezione, esploreremo la struttura dati delle liste-colonne un po’ più in dettaglio. È solo di recente che ho veramente apprezzato l’idea della colonna-elenco. Le liste-colonne sono implicite nella definizione del data frame: un data frame è una lista nominata di vettori di uguale lunghezza. Una lista è un vettore, quindi è sempre stato legittimo usare una lista come colonna di un data frame. Tuttavia, R di base non rende facile la creazione di liste-colonne, e data.frame() tratta una lista come una lista di colonne:.
data.frame(x = list(1:3, 3:5))
#> x.1.3 x.3.5
#> 1 1 3
#> 2 2 4
#> 3 3 5Potete evitare che data.frame() faccia questo con I(), ma il risultato non viene stampato particolarmente bene:
data.frame(
x = I(list(1:3, 3:5)),
y = c("1, 2", "3, 4, 5")
)
#> x y
#> 1 1, 2, 3 1, 2
#> 2 3, 4, 5 3, 4, 5Tibble allevia questo problema essendo più pigro (tibble() non modifica i suoi input) e fornendo un metodo di stampa migliore:
tibble(
x = list(1:3, 3:5),
y = c("1, 2", "3, 4, 5")
)
#> # A tibble: 2 × 2
#> x y
#> <list> <chr>
#> 1 <int [3]> 1, 2
#> 2 <int [3]> 3, 4, 5È ancora più facile con tribble() in quanto può capire automaticamente che avete bisogno di una lista:
tribble(
~x, ~y,
1:3, "1, 2",
3:5, "3, 4, 5"
)
#> # A tibble: 2 × 2
#> x y
#> <list> <chr>
#> 1 <int [3]> 1, 2
#> 2 <int [3]> 3, 4, 5Le colonne-elenco sono spesso molto utili come struttura dati intermedia. Sono difficili da lavorare direttamente, perché la maggior parte delle funzioni di R lavora con vettori atomici o frame di dati, ma il vantaggio di tenere insieme elementi correlati in un frame di dati vale un po’ di fastidio.
Generalmente ci sono tre parti di una efficace pipeline lista-colonna:
Si crea la lista-colonna usando uno dei metodi
nest(),summarise()+list(), omutate()+ una funzione di mappa, come descritto in [Creare liste-colonne].Si creano altre colonne-elenco intermedie trasformando le colonne-elenco esistenti con colonne della lista con
map(),map2()opmap(). Per esempio, nel caso di studio precedente, abbiamo creato una lista-colonna di modelli trasformando una colonna-elenco di frame di dati.Si semplifica la lista-colonna riducendola a un data frame o a un vettore atomico, come descritto in [Simplifying list-columns].
25.4 Creazione di list-columns
Tipicamente, non si creano colonne-elenco con tibble(). Invece, le creerai da colonne regolari, usando uno dei tre metodi:
Con
tidyr::nest()per convertire un data frame raggruppato in un data frame annidato in un frame di dati annidato in cui si ha una lista-colonna di frame di dati.- Con
mutate()e funzioni vettoriali che restituiscono una lista.
- Con
Con
summarise()e le funzioni di riepilogo che restituiscono più risultati.
In alternativa, potreste crearli da una lista nominata, usando tibble::enframe().
Generalmente, quando si creano colonne di liste, ci si dovrebbe assicurare che siano omogenee: ogni elemento dovrebbe contenere lo stesso tipo di cose. Non ci sono controlli per assicurarsi che questo sia vero, ma se usate purrr e ricordate ciò che avete imparato sulle funzioni type-stable, dovreste scoprire che ciò avviene naturalmente.
25.4.1 Con l’annidamento
nest() crea un data frame annidato, che è un data frame con una lista-colonna di data frame. In un data frame annidato ogni riga è una meta-osservazione: le altre colonne danno le variabili che definiscono l’osservazione (come il paese e il continente sopra), e la lista-colonna di data frame dà le singole osservazioni che compongono la meta-osservazione.
Ci sono due modi per usare nest(). Finora avete visto come usarlo con un frame di dati raggruppato. Quando viene applicato ad un frame di dati raggruppati, nest() mantiene le colonne di raggruppamento così come sono, e raggruppa tutto il resto nella lista-colonna:
gapminder %>%
group_by(country, continent) %>%
nest()
#> # A tibble: 142 × 3
#> # Groups: country, continent [142]
#> country continent data
#> <fct> <fct> <list>
#> 1 Afghanistan Asia <tibble [12 × 4]>
#> 2 Albania Europe <tibble [12 × 4]>
#> 3 Algeria Africa <tibble [12 × 4]>
#> 4 Angola Africa <tibble [12 × 4]>
#> 5 Argentina Americas <tibble [12 × 4]>
#> 6 Australia Oceania <tibble [12 × 4]>
#> # … with 136 more rowsPotete anche usarlo su un frame di dati non raggruppato, specificando quali colonne volete annidare:
gapminder %>%
nest(data = c(year:gdpPercap))
#> # A tibble: 142 × 3
#> country continent data
#> <fct> <fct> <list>
#> 1 Afghanistan Asia <tibble [12 × 4]>
#> 2 Albania Europe <tibble [12 × 4]>
#> 3 Algeria Africa <tibble [12 × 4]>
#> 4 Angola Africa <tibble [12 × 4]>
#> 5 Argentina Americas <tibble [12 × 4]>
#> 6 Australia Oceania <tibble [12 × 4]>
#> # … with 136 more rows25.4.2 Dalle funzioni vettoriali
Alcune funzioni utili prendono un vettore atomico e restituiscono una lista. Per esempio, in [strings] avete imparato a conoscere stringr::str_split() che prende un vettore di caratteri e restituisce una lista di vettori di caratteri. Se lo usi dentro mutate, otterrai una lista-colonna:
df <- tribble(
~x1,
"a,b,c",
"d,e,f,g"
)
df %>%
mutate(x2 = stringr::str_split(x1, ","))
#> # A tibble: 2 × 2
#> x1 x2
#> <chr> <list>
#> 1 a,b,c <chr [3]>
#> 2 d,e,f,g <chr [4]>unnest() sa come gestire queste liste di vettori:
df %>%
mutate(x2 = stringr::str_split(x1, ",")) %>%
unnest(x2)
#> # A tibble: 7 × 2
#> x1 x2
#> <chr> <chr>
#> 1 a,b,c a
#> 2 a,b,c b
#> 3 a,b,c c
#> 4 d,e,f,g d
#> 5 d,e,f,g e
#> 6 d,e,f,g f
#> # … with 1 more row(Se vi trovate ad usare spesso questo schema, assicuratevi di controllare tidyr::separate_rows() che è un wrapper intorno a questo schema comune).
Un altro esempio di questo pattern è l’uso di map(), map2(), pmap() da purrr. Per esempio, potremmo prendere l’esempio finale da [Invoking different functions] e riscriverlo per usare mutate():
sim <- tribble(
~f, ~params,
"runif", list(min = -1, max = 1),
"rnorm", list(sd = 5),
"rpois", list(lambda = 10)
)
sim %>%
mutate(sims = invoke_map(f, params, n = 10))
#> Warning: There was 1 warning in `mutate()`.
#> ℹ In argument: `sims = invoke_map(f, params, n = 10)`.
#> Caused by warning:
#> ! `invoke_map()` was deprecated in purrr 1.0.0.
#> ℹ Please use map() + exec() instead.
#> # A tibble: 3 × 3
#> f params sims
#> <chr> <list> <list>
#> 1 runif <named list [2]> <dbl [10]>
#> 2 rnorm <named list [1]> <dbl [10]>
#> 3 rpois <named list [1]> <int [10]>Notate che tecnicamente sim non è omogeneo perché contiene sia vettori doppi che interi. Tuttavia, è improbabile che questo causi molti problemi, dato che interi e doppi sono entrambi vettori numerici.
25.4.3 Dai sommari multivalutati
Una restrizione di summarise() è che funziona solo con funzioni di riepilogo che restituiscono un singolo valore. Ciò significa che non potete usarla con funzioni come quantile() che restituiscono un vettore di lunghezza arbitraria:
mtcars %>%
group_by(cyl) %>%
summarise(q = quantile(mpg))
#> Warning: Returning more (or less) than 1 row per `summarise()` group was deprecated in
#> dplyr 1.1.0.
#> ℹ Please use `reframe()` instead.
#> ℹ When switching from `summarise()` to `reframe()`, remember that `reframe()`
#> always returns an ungrouped data frame and adjust accordingly.
#> `summarise()` has grouped output by 'cyl'. You can override using the `.groups`
#> argument.
#> # A tibble: 15 × 2
#> # Groups: cyl [3]
#> cyl q
#> <dbl> <dbl>
#> 1 4 21.4
#> 2 4 22.8
#> 3 4 26
#> 4 4 30.4
#> 5 4 33.9
#> 6 6 17.8
#> # … with 9 more rowsPotete però avvolgere il risultato in una lista! Questo obbedisce al contratto di summarise(), perché ogni sommario è ora una lista (un vettore) di lunghezza 1.
mtcars %>%
group_by(cyl) %>%
summarise(q = list(quantile(mpg)))
#> # A tibble: 3 × 2
#> cyl q
#> <dbl> <list>
#> 1 4 <dbl [5]>
#> 2 6 <dbl [5]>
#> 3 8 <dbl [5]>Per ottenere risultati utili con unnest, dovrete anche catturare le probabilità:
probs <- c(0.01, 0.25, 0.5, 0.75, 0.99)
mtcars %>%
group_by(cyl) %>%
summarise(p = list(probs), q = list(quantile(mpg, probs))) %>%
unnest(c(p, q))
#> # A tibble: 15 × 3
#> cyl p q
#> <dbl> <dbl> <dbl>
#> 1 4 0.01 21.4
#> 2 4 0.25 22.8
#> 3 4 0.5 26
#> 4 4 0.75 30.4
#> 5 4 0.99 33.8
#> 6 6 0.01 17.8
#> # … with 9 more rows25.4.4 Da un elenco con nome
I data frame con colonne di liste forniscono una soluzione ad un problema comune: cosa fare se si vuole iterare sia il contenuto di una lista che i suoi elementi? Invece di cercare di incastrare tutto in un solo oggetto, è spesso più facile fare un data frame: una colonna può contenere gli elementi, e una colonna può contenere la lista. Un modo semplice per creare un tale data frame da una lista è tibble::enframe().
x <- list(
a = 1:5,
b = 3:4,
c = 5:6
)
df <- enframe(x)
df
#> # A tibble: 3 × 2
#> name value
#> <chr> <list>
#> 1 a <int [5]>
#> 2 b <int [2]>
#> 3 c <int [2]>Il vantaggio di questa struttura è che si generalizza in modo diretto - i nomi sono utili se avete un vettore di metadati a carattere, ma non aiutano se avete altri tipi di dati, o vettori multipli.
Ora, se volete iterare su nomi e valori in parallelo, potete usare map2():
25.4.5 Esercizi
Elenca tutte le funzioni che ti vengono in mente che prendono un vettore atomico e restituire una lista.
Inventa utili funzioni di riepilogo che, come la
quantile(), restituiscono valori multipli.-
Cosa manca nel seguente data frame? Come fa la funzione
quantile()a restituire quel pezzo mancante? Perché non è utile in questo caso? -
Cosa fa questo codice? Perché potrebbe essere utile?
mtcars %>% group_by(cyl) %>% summarise_all(list(list))
25.5 Semplificare le list-columns
Per applicare le tecniche di manipolazione e visualizzazione dei dati che hai imparato in questo libro, avrai bisogno di semplificare la lista-colonna a una colonna regolare (un vettore atomico), o un insieme di colonne. La tecnica che userete per ridurla ad una struttura più semplice dipende dal fatto che vogliate un singolo valore per elemento o più valori:
Se volete un singolo valore, usate
mutate()conmap_lgl(),map_int(),map_dbl(), emap_chr()per creare un vettore atomico.Se volete molti valori, usate
unnest()per convertire le colonne della lista a colonne regolari, ripetendo le righe tante volte quanto necessario.
Questi sono descritti più in dettaglio qui di seguito.
25.5.1 Da lista a vettore
Se si può ridurre la colonna della lista ad un vettore atomico, allora sarà una colonna regolare. Per esempio, potete sempre riassumere un oggetto con il suo tipo e la sua lunghezza, quindi questo codice funzionerà indipendentemente dal tipo di colonna elenco che avete:
df <- tribble(
~x,
letters[1:5],
1:3,
runif(5)
)
df %>% mutate(
type = map_chr(x, typeof),
length = map_int(x, length)
)
#> # A tibble: 3 × 3
#> x type length
#> <list> <chr> <int>
#> 1 <chr [5]> character 5
#> 2 <int [3]> integer 3
#> 3 <dbl [5]> double 5Queste sono le stesse informazioni di base che si ottengono dal metodo di stampa predefinito di tbl, ma ora si possono usare per filtrare. Questa è una tecnica utile se avete una lista eterogenea e volete filtrare le parti che non funzionano.
Non dimenticate le scorciatoie map_*() - potete usare map_chr(x, "apple") per estrarre la stringa memorizzata in apple per ogni elemento di x. Questo è utile per estrarre liste annidate in colonne regolari. Usate l’argomento .null per fornire un valore da usare se l’elemento manca (invece di restituire NULL):
25.5.2 Unnesting
unnest() funziona ripetendo le colonne regolari una volta per ogni elemento della lista-colonna. Per esempio, nel seguente esempio molto semplice ripetiamo la prima riga 4 volte (perché lì il primo elemento di y ha lunghezza quattro), e la seconda riga una volta:
tibble(x = 1:2, y = list(1:4, 1)) %>% unnest(y)
#> # A tibble: 5 × 2
#> x y
#> <int> <dbl>
#> 1 1 1
#> 2 1 2
#> 3 1 3
#> 4 1 4
#> 5 2 1Questo significa che non si può contemporaneamente snidare due colonne che contengono un numero diverso di elementi:
# Ok, perché y e z hanno lo stesso numero di elementi in
# ogni riga
df1 <- tribble(
~x, ~y, ~z,
1, c("a", "b"), 1:2,
2, "c", 3
)
df1
#> # A tibble: 2 × 3
#> x y z
#> <dbl> <list> <list>
#> 1 1 <chr [2]> <int [2]>
#> 2 2 <chr [1]> <dbl [1]>
df1 %>% unnest(c(y, z))
#> # A tibble: 3 × 3
#> x y z
#> <dbl> <chr> <dbl>
#> 1 1 a 1
#> 2 1 b 2
#> 3 2 c 3
# Non funziona perché y e z hanno un numero diverso di elementi
df2 <- tribble(
~x, ~y, ~z,
1, "a", 1:2,
2, c("b", "c"), 3
)
df2
#> # A tibble: 2 × 3
#> x y z
#> <dbl> <list> <list>
#> 1 1 <chr [1]> <int [2]>
#> 2 2 <chr [2]> <dbl [1]>
df2 %>% unnest(c(y, z))
#> # A tibble: 4 × 3
#> x y z
#> <dbl> <chr> <dbl>
#> 1 1 a 1
#> 2 1 a 2
#> 3 2 b 3
#> 4 2 c 3Lo stesso principio si applica quando si unnestano le colonne dell’elenco dei frame di dati. Potete unnestare più colonne-elenco finché tutti i frame di dati in ogni riga hanno lo stesso numero di righe.
25.5.3 Esercizi
Perché la funzione
lengths()potrebbe essere utile per creare colonne colonne vettoriali da colonne-elenco?Elencare i tipi più comuni di vettore che si trovano in un data frame. Cosa rende diverse le liste?
25.6 Rendere ordinati i dati con broom
Il pacchetto broom fornisce tre strumenti generali per trasformare i modelli in frame di dati ordinati:
broom::glance(model)restituisce una riga per ogni modello. Ogni colonna fornisce un riepilogo del modello: o una misura della qualità del modello, o della complessità, o una combinazione dei due.-
broom::tidy(model)restituisce una riga per ogni coefficiente nel modello. Ogni colonna fornisce informazioni sulla stima o sulla sua variabilità.
-
-
broom::augment(model, data)restituisce una riga per ogni riga indata, aggiungendo valori extra come i residui e le statistiche di influenza.
-